Attention: World-2DPAGE is no longer maintained.
[Click to access protein entry]
Spot: 2D-001KKJ (arabidopsis)
pI: 5.32 Mw: 30434
%vol: 0.231462 %od: 0.207945
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KKJ (arabidopsis)
pI: 5.32 Mw: 30434
%vol: 0.231462 %od: 0.207945
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001K3R (arabidopsis)
pI: 4.71 Mw: 58163
%vol: 0.276269 %od: 0.234557
================
*PDI11_ARATH*
accession n°: Q9XI01
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 972.508 (0), 1033.481 (0), 1034.484 (0), 1352.679 (0),
1589.835 (0), 1599.776 (0), 1796.865 (0) }
Spot: 2D-001K3R (arabidopsis)
pI: 4.71 Mw: 58163
%vol: 0.276269 %od: 0.234557
================
*PDI11_ARATH*
accession n°: Q9XI01
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 972.508 (0), 1033.481 (0), 1034.484 (0), 1352.679 (0),
1589.835 (0), 1599.776 (0), 1796.865 (0) }
[Click to access protein entry]
Spot: 2D-001KM4 (arabidopsis)
pI: 5.43 Mw: 28620
%vol: 0.210881 %od: 0.175446
================
*CAHC_ARATH*
accession n°: P27140
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 948.408 (0), 1251.513 (0), 1261.61 (0), 1468.7 (0), 1725.88 (0),
1782.841 (0), 1906.093 (0), 2043.055 (0) }
Spot: 2D-001KM4 (arabidopsis)
pI: 5.43 Mw: 28620
%vol: 0.210881 %od: 0.175446
================
*CAHC_ARATH*
accession n°: P27140
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 948.408 (0), 1251.513 (0), 1261.61 (0), 1468.7 (0), 1725.88 (0),
1782.841 (0), 1906.093 (0), 2043.055 (0) }
[Click to access protein entry]
Spot: 2D-001KOU (arabidopsis)
pI: 5.26 Mw: 21689
%vol: 0.048194 %od: 0.0849044
================
*CP20C_ARATH*
accession n°: P34791
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 963.44 (0), 1142.53 (0), 1304.735 (0), 1510.797 (0)
}
Spot: 2D-001KOU (arabidopsis)
pI: 5.26 Mw: 21689
%vol: 0.048194 %od: 0.0849044
================
*CP20C_ARATH*
accession n°: P34791
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 963.44 (0), 1142.53 (0), 1304.735 (0), 1510.797 (0)
}
[Click to access protein entry]
Spot: 2D-001KLM (arabidopsis)
pI: 5.36 Mw: 29087
%vol: 0.270408 %od: 0.432962
================
*TPIS_ARATH*
accession n°: P48491
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KLM (arabidopsis)
pI: 5.36 Mw: 29087
%vol: 0.270408 %od: 0.432962
================
*TPIS_ARATH*
accession n°: P48491
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KLZ (arabidopsis)
pI: 5.71 Mw: 28620
%vol: 0.336186 %od: 0.772182
================
*CAHC_ARATH*
accession n°: P27140
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 948.428 (0), 1019.514 (0), 1135.573 (0), 1251.523 (0),
1261.618 (0), 1468.744 (0), 1652.912 (0), 1660.924 (0), 1725.88 (0),
1782.833 (0), 1888.093 (0), 1906.089 (0), 1986.036 (0), 2043.07 (0)
}
Spot: 2D-001KLZ (arabidopsis)
pI: 5.71 Mw: 28620
%vol: 0.336186 %od: 0.772182
================
*CAHC_ARATH*
accession n°: P27140
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 948.428 (0), 1019.514 (0), 1135.573 (0), 1251.523 (0),
1261.618 (0), 1468.744 (0), 1652.912 (0), 1660.924 (0), 1725.88 (0),
1782.833 (0), 1888.093 (0), 1906.089 (0), 1986.036 (0), 2043.07 (0)
}
[Click to access protein entry]
Spot: 2D-001UMS (arabidopsis)
pI: 6.08 Mw: 14843
%vol: 0.361195 %od: 1.54601
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {Gm} {PMF}
Spot: 2D-001UMS (arabidopsis)
pI: 6.08 Mw: 14843
%vol: 0.361195 %od: 1.54601
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {Gm} {PMF}
[Click to access protein entry]
Spot: 2D-001KJO (arabidopsis)
pI: 5.00 Mw: 31181
%vol: 0.280437 %od: 0.49428
================
*PSBO1_ARATH*
accession n°: P23321
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.443 (0), 950.528 (0), 1252.642 (0), 1362.67 (0),
1433.746 (0), 1490.763 (0), 1562.781 (0), 1646.927 (0), 2604.423 (0)
}
================
*PSBO2_ARATH*
accession n: Q9S841
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.384 (0), 950.511 (0), 1252.568 (0), 1562.685 (0),
2174.815 (0), 2255.968 (0), 2294.037 (0), 2604.181 (0)
}
Spot: 2D-001KJO (arabidopsis)
pI: 5.00 Mw: 31181
%vol: 0.280437 %od: 0.49428
================
*PSBO1_ARATH*
accession n°: P23321
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.443 (0), 950.528 (0), 1252.642 (0), 1362.67 (0),
1433.746 (0), 1490.763 (0), 1562.781 (0), 1646.927 (0), 2604.423 (0)
}
================
*PSBO2_ARATH*
accession n: Q9S841
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.384 (0), 950.511 (0), 1252.568 (0), 1562.685 (0),
2174.815 (0), 2255.968 (0), 2294.037 (0), 2604.181 (0)
}
[Click to access protein entry]
Spot: 2D-001KKP (arabidopsis)
pI: 5.55 Mw: 30287
%vol: 0.0557489 %od: 0.0349507
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.416 (0), 1021.517 (0), 1407.685 (0), 1465.778 (0),
1475.774 (0), 1502.883 (0), 2186.073 (0) }
Spot: 2D-001KKP (arabidopsis)
pI: 5.55 Mw: 30287
%vol: 0.0557489 %od: 0.0349507
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.416 (0), 1021.517 (0), 1407.685 (0), 1465.778 (0),
1475.774 (0), 1502.883 (0), 2186.073 (0) }
[Click to access protein entry]
Spot: 2D-001K6G (arabidopsis)
pI: 5.35 Mw: 52568
%vol: 0.325635 %od: 0.413749
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 975.509 (0), 1007.545 (0), 1045.564 (0), 1201.652 (0),
1328.699 (0), 1415.743 (0), 1433.787 (0), 1617.807 (0), 1630.852 (0),
1955.066 (0) }
Spot: 2D-001K6G (arabidopsis)
pI: 5.35 Mw: 52568
%vol: 0.325635 %od: 0.413749
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 975.509 (0), 1007.545 (0), 1045.564 (0), 1201.652 (0),
1328.699 (0), 1415.743 (0), 1433.787 (0), 1617.807 (0), 1630.852 (0),
1955.066 (0) }
[Click to access protein entry]
Spot: 2D-001KE8 (arabidopsis)
pI: 6.58 Mw: 39317
%vol: 0.314954 %od: 0.359652
================
*S17P_ARATH*
accession n°: P46283
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 913.471 (0), 937.444 (0), 959.486 (0), 1161.498 (0),
1202.572 (0), 1235.639 (0), 1607.82 (0), 1649.868 (0), 1659.851 (0),
1963.968 (0) }
Spot: 2D-001KE8 (arabidopsis)
pI: 6.58 Mw: 39317
%vol: 0.314954 %od: 0.359652
================
*S17P_ARATH*
accession n°: P46283
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 913.471 (0), 937.444 (0), 959.486 (0), 1161.498 (0),
1202.572 (0), 1235.639 (0), 1607.82 (0), 1649.868 (0), 1659.851 (0),
1963.968 (0) }
[Click to access protein entry]
Spot: 2D-001K9S (arabidopsis)
pI: 6.15 Mw: 47034
%vol: 0.0703371 %od: 0.0345201
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.489 (0), 914.439 (0), 1021.563 (0), 1249.681 (0),
1465.702 (0), 1502.784 (0) }
Spot: 2D-001K9S (arabidopsis)
pI: 6.15 Mw: 47034
%vol: 0.0703371 %od: 0.0345201
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.489 (0), 914.439 (0), 1021.563 (0), 1249.681 (0),
1465.702 (0), 1502.784 (0) }
[Click to access protein entry]
Spot: 2D-001KLQ (arabidopsis)
pI: 5.22 Mw: 28946
%vol: 0.101077 %od: 0.0909185
================
*TPIS_ARATH*
accession n°: P48491
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KLQ (arabidopsis)
pI: 5.22 Mw: 28946
%vol: 0.101077 %od: 0.0909185
================
*TPIS_ARATH*
accession n°: P48491
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001K7F (arabidopsis)
pI: 5.29 Mw: 50968
%vol: 0.332409 %od: 1.2811
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 975.238 (0), 1007.293 (0), 1045.237 (0), 1191.188 (0),
1277.275 (0), 1328.242 (0), 1415.307 (0), 1433.362 (0), 1617.319 (0),
1630.308 (0), 1734.492 (0), 1949.376 (0), 1954.463 (0), 2076.418 (0),
2173.465 (0), 2327.449 (0), 2400.459 (0), 2461.507 (0), 2613.555 (0)
}
Spot: 2D-001K7F (arabidopsis)
pI: 5.29 Mw: 50968
%vol: 0.332409 %od: 1.2811
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 975.238 (0), 1007.293 (0), 1045.237 (0), 1191.188 (0),
1277.275 (0), 1328.242 (0), 1415.307 (0), 1433.362 (0), 1617.319 (0),
1630.308 (0), 1734.492 (0), 1949.376 (0), 1954.463 (0), 2076.418 (0),
2173.465 (0), 2327.449 (0), 2400.459 (0), 2461.507 (0), 2613.555 (0)
}
[Click to access protein entry]
Spot: 2D-001K9W (arabidopsis)
pI: 6.23 Mw: 46937
%vol: 0.163208 %od: 0.143982
================
*MB23_ARATH*
accession n°: O80948
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 913.527 (0), 966.43 (0), 1063.551 (0), 1094.544 (0),
1191.641 (0), 1240.51 (0), 1267.618 (0), 1334.66 (0), 1382.595 (0),
1438.789 (0), 1540.774 (0), 1595.818 (0), 1744.008 (0), 1991.954 (0),
2292.115 (0) }
Spot: 2D-001K9W (arabidopsis)
pI: 6.23 Mw: 46937
%vol: 0.163208 %od: 0.143982
================
*MB23_ARATH*
accession n°: O80948
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 913.527 (0), 966.43 (0), 1063.551 (0), 1094.544 (0),
1191.641 (0), 1240.51 (0), 1267.618 (0), 1334.66 (0), 1382.595 (0),
1438.789 (0), 1540.774 (0), 1595.818 (0), 1744.008 (0), 1991.954 (0),
2292.115 (0) }
[Click to access protein entry]
Spot: 2D-001K6F (arabidopsis)
pI: 5.29 Mw: 52676
%vol: 0.21518 %od: 0.17777
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.402 (0), 1201.452 (0), 1263.507 (0), 1328.429 (0),
1433.555 (0), 1617.524 (0), 1630.514 (0), 1949.715 (0), 1954.802 (0),
2327.819 (0), 2841.981 (0) }
Spot: 2D-001K6F (arabidopsis)
pI: 5.29 Mw: 52676
%vol: 0.21518 %od: 0.17777
================
*ATPB_ARATH*
accession n°: P19366
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.402 (0), 1201.452 (0), 1263.507 (0), 1328.429 (0),
1433.555 (0), 1617.524 (0), 1630.514 (0), 1949.715 (0), 1954.802 (0),
2327.819 (0), 2841.981 (0) }
[Click to access protein entry]
Spot: 2D-001KAH (arabidopsis)
pI: 4.96 Mw: 45509
%vol: 0.0945642 %od: 0.0605917
================
*METK1_ARATH*
accession n°: P99056
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 979.481 (0), 1141.573 (0), 1453.682 (0), 2377.975 (0)
}
Spot: 2D-001KAH (arabidopsis)
pI: 4.96 Mw: 45509
%vol: 0.0945642 %od: 0.0605917
================
*METK1_ARATH*
accession n°: P99056
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 979.481 (0), 1141.573 (0), 1453.682 (0), 2377.975 (0)
}
[Click to access protein entry]
Spot: 2D-001JZ9 (arabidopsis)
pI: 5.08 Mw: 98398
%vol: 0.272622 %od: 0.278404
================
*LOX2_ARATH*
accession n°: P38418
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 919.491 (0), 958.474 (0), 987.442 (0), 1064.611 (0),
1087.506 (0), 1094.526 (0), 1208.576 (0), 1231.583 (0), 1281.61 (0),
1334.679 (0), 1345.652 (0), 1487.735 (0), 1599.809 (0), 1616.732 (0),
1716.819 (0), 1962.893 (0), 2198.067 (0) }
Spot: 2D-001JZ9 (arabidopsis)
pI: 5.08 Mw: 98398
%vol: 0.272622 %od: 0.278404
================
*LOX2_ARATH*
accession n°: P38418
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 919.491 (0), 958.474 (0), 987.442 (0), 1064.611 (0),
1087.506 (0), 1094.526 (0), 1208.576 (0), 1231.583 (0), 1281.61 (0),
1334.679 (0), 1345.652 (0), 1487.735 (0), 1599.809 (0), 1616.732 (0),
1716.819 (0), 1962.893 (0), 2198.067 (0) }
[Click to access protein entry]
Spot: 2D-001K0N (arabidopsis)
pI: 6.15 Mw: 76088
%vol: 0.262202 %od: 0.170323
================
*METE_ARATH*
accession n°: O50008
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001K0N (arabidopsis)
pI: 6.15 Mw: 76088
%vol: 0.262202 %od: 0.170323
================
*METE_ARATH*
accession n°: O50008
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KL4 (arabidopsis)
pI: 5.15 Mw: 29657
%vol: 0.0788036 %od: 0.0645598
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KL4 (arabidopsis)
pI: 5.15 Mw: 29657
%vol: 0.0788036 %od: 0.0645598
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KLL (arabidopsis)
pI: 5.73 Mw: 29134
%vol: 0.246832 %od: 0.294586
================
*APX1_ARATH*
accession n°: Q05431
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 939.411 (0), 976.439 (0), 1249.609 (0), 1309.696 (0),
1343.647 (0), 1611.918 (0), 1635.752 (0), 1906.961 (0), 2080.877 (0),
2085.063 (0) }
Spot: 2D-001KLL (arabidopsis)
pI: 5.73 Mw: 29134
%vol: 0.246832 %od: 0.294586
================
*APX1_ARATH*
accession n°: Q05431
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 939.411 (0), 976.439 (0), 1249.609 (0), 1309.696 (0),
1343.647 (0), 1611.918 (0), 1635.752 (0), 1906.961 (0), 2080.877 (0),
2085.063 (0) }
[Click to access protein entry]
Spot: 2D-001KMI (arabidopsis)
pI: 5.86 Mw: 28024
%vol: 0.232113 %od: 0.25965
================
*GSTF2_ARATH*
accession n°: P46422
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KMI (arabidopsis)
pI: 5.86 Mw: 28024
%vol: 0.232113 %od: 0.25965
================
*GSTF2_ARATH*
accession n°: P46422
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KBJ (arabidopsis)
pI: 5.34 Mw: 43943
%vol: 0.254517 %od: 0.131149
================
*ACT8_ARATH*
accession n°: P99055
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.443 (0), 1008.624 (0), 1184.672 (0), 1192.51 (0),
1338.642 (0), 1413.666 (0), 1774.892 (0), 1968.051 (0)
}
Spot: 2D-001KBJ (arabidopsis)
pI: 5.34 Mw: 43943
%vol: 0.254517 %od: 0.131149
================
*ACT8_ARATH*
accession n°: P99055
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.443 (0), 1008.624 (0), 1184.672 (0), 1192.51 (0),
1338.642 (0), 1413.666 (0), 1774.892 (0), 1968.051 (0)
}
[Click to access protein entry]
Spot: 2D-001K6U (arabidopsis)
pI: 5.45 Mw: 52029
%vol: 0.31287 %od: 0.313242
================
*ENO2_ARATH*
accession n°: P25696
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001K6U (arabidopsis)
pI: 5.45 Mw: 52029
%vol: 0.31287 %od: 0.313242
================
*ENO2_ARATH*
accession n°: P25696
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KQE (arabidopsis)
pI: 5.79 Mw: 17718
%vol: 0.180662 %od: 0.310315
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.473 (0), 935.506 (0), 952.484 (0), 1080.556 (0),
1365.556 (0), 1548.76 (0), 1939.94 (0), 2068.072 (0), 2086.026 (0)
}
Spot: 2D-001KQE (arabidopsis)
pI: 5.79 Mw: 17718
%vol: 0.180662 %od: 0.310315
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.473 (0), 935.506 (0), 952.484 (0), 1080.556 (0),
1365.556 (0), 1548.76 (0), 1939.94 (0), 2068.072 (0), 2086.026 (0)
}
[Click to access protein entry]
Spot: 2D-001KC0 (arabidopsis)
pI: 5.23 Mw: 42958
%vol: 0.200201 %od: 0.190336
================
*G3PB_ARATH*
accession n°: P25857
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1067.654 (0), 1077.463 (0), 1148.599 (0), 1151.666 (0),
1289.624 (0), 1321.636 (0), 1449.734 (0), 1571.668 (0), 1627.831 (0),
1755.925 (0), 1772.798 (0), 1775.87 (0) }
Spot: 2D-001KC0 (arabidopsis)
pI: 5.23 Mw: 42958
%vol: 0.200201 %od: 0.190336
================
*G3PB_ARATH*
accession n°: P25857
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1067.654 (0), 1077.463 (0), 1148.599 (0), 1151.666 (0),
1289.624 (0), 1321.636 (0), 1449.734 (0), 1571.668 (0), 1627.831 (0),
1755.925 (0), 1772.798 (0), 1775.87 (0) }
[Click to access protein entry]
Spot: 2D-001KKI (arabidopsis)
pI: 5.22 Mw: 30385
%vol: 0.197335 %od: 0.140413
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KKI (arabidopsis)
pI: 5.22 Mw: 30385
%vol: 0.197335 %od: 0.140413
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001K7A (arabidopsis)
pI: 5.78 Mw: 51284
%vol: 0.318602 %od: 0.973722
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.458 (0), 914.405 (0), 928.455 (0), 962.47 (0), 1021.537 (0),
1039.468 (0), 1116.604 (0), 1187.667 (0), 1228.675 (0), 1249.698 (0),
1261.63 (0), 1324.711 (0), 1447.779 (0), 1465.767 (0), 1502.873 (0),
1546.793 (0), 1819.934 (0), 1966.92 (0) }
Spot: 2D-001K7A (arabidopsis)
pI: 5.78 Mw: 51284
%vol: 0.318602 %od: 0.973722
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.458 (0), 914.405 (0), 928.455 (0), 962.47 (0), 1021.537 (0),
1039.468 (0), 1116.604 (0), 1187.667 (0), 1228.675 (0), 1249.698 (0),
1261.63 (0), 1324.711 (0), 1447.779 (0), 1465.767 (0), 1502.873 (0),
1546.793 (0), 1819.934 (0), 1966.92 (0) }
[Click to access protein entry]
Spot: 2D-001KQA (arabidopsis)
pI: 6.19 Mw: 19999
%vol: 0.067211 %od: 0.0619313
================
*NDK1_ARATH*
accession n°: P39207
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KQA (arabidopsis)
pI: 6.19 Mw: 19999
%vol: 0.067211 %od: 0.0619313
================
*NDK1_ARATH*
accession n°: P39207
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KLH (arabidopsis)
pI: 4.66 Mw: 29181
%vol: 0.0631732 %od: 0.0573918
================
*ROC1_ARATH*
accession n°: Q9ZUU4
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1018.459 (0), 1024.489 (0), 1111.617 (0), 1267.71 (0),
1906.832 (0) }
Spot: 2D-001KLH (arabidopsis)
pI: 4.66 Mw: 29181
%vol: 0.0631732 %od: 0.0573918
================
*ROC1_ARATH*
accession n°: Q9ZUU4
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1018.459 (0), 1024.489 (0), 1111.617 (0), 1267.71 (0),
1906.832 (0) }
[Click to access protein entry]
Spot: 2D-001KDM (arabidopsis)
pI: 5.55 Mw: 40467
%vol: 0.0507991 %od: 0.0283146
================
*ODPA1_ARATH*
accession n°: P52901
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 922.47 (0), 1017.482 (0), 1169.544 (0), 1297.629 (0),
1307.682 (0), 1323.666 (0), 1534.726 (0), 1610.699 (0), 1643.797 (0)
}
Spot: 2D-001KDM (arabidopsis)
pI: 5.55 Mw: 40467
%vol: 0.0507991 %od: 0.0283146
================
*ODPA1_ARATH*
accession n°: P52901
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 922.47 (0), 1017.482 (0), 1169.544 (0), 1297.629 (0),
1307.682 (0), 1323.666 (0), 1534.726 (0), 1610.699 (0), 1643.797 (0)
}
[Click to access protein entry]
Spot: 2D-001K6O (arabidopsis)
pI: 5.71 Mw: 50864
%vol: 0.336967 %od: 3.58771
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {Gm} {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 898.518 (0), 910.532 (0), 914.49 (0), 928.539 (0), 962.545 (0),
1021.609 (0), 1039.55 (0), 1059.631 (0), 1116.649 (0), 1187.73 (0),
1228.715 (0), 1249.739 (0), 1261.664 (0), 1324.731 (0), 1407.697 (0),
1447.818 (0), 1451.666 (0), 1465.803 (0), 1502.902 (0), 1532.766 (0),
1546.758 (0), 1810.815 (0), 1819.963 (0), 1866.904 (0), 1966.889 (0),
1998.962 (0), 2185.925 (0) }
Spot: 2D-001K6O (arabidopsis)
pI: 5.71 Mw: 50864
%vol: 0.336967 %od: 3.58771
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {Gm} {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 898.518 (0), 910.532 (0), 914.49 (0), 928.539 (0), 962.545 (0),
1021.609 (0), 1039.55 (0), 1059.631 (0), 1116.649 (0), 1187.73 (0),
1228.715 (0), 1249.739 (0), 1261.664 (0), 1324.731 (0), 1407.697 (0),
1447.818 (0), 1451.666 (0), 1465.803 (0), 1502.902 (0), 1532.766 (0),
1546.758 (0), 1810.815 (0), 1819.963 (0), 1866.904 (0), 1966.889 (0),
1998.962 (0), 2185.925 (0) }
[Click to access protein entry]
Spot: 2D-001K45 (arabidopsis)
pI: 4.81 Mw: 57331
%vol: 0.207234 %od: 0.12427
================
*CPNA1_ARATH*
accession n°: P21238
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1062.584 (0), 1102.54 (0), 1290.633 (0), 1479.768 (0),
1561.716 (0), 1736.009 (0), 1904.972 (0) }
Spot: 2D-001K45 (arabidopsis)
pI: 4.81 Mw: 57331
%vol: 0.207234 %od: 0.12427
================
*CPNA1_ARATH*
accession n°: P21238
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1062.584 (0), 1102.54 (0), 1290.633 (0), 1479.768 (0),
1561.716 (0), 1736.009 (0), 1904.972 (0) }
[Click to access protein entry]
Spot: 2D-001KMQ (arabidopsis)
pI: 6.30 Mw: 27754
%vol: 0.108111 %od: 0.122599
================
*GSTF7_ARATH*
accession n°: Q9SRY5
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 936.507 (0), 1030.521 (0), 1124.528 (0), 1143.599 (0),
1503.818 (0) }
Spot: 2D-001KMQ (arabidopsis)
pI: 6.30 Mw: 27754
%vol: 0.108111 %od: 0.122599
================
*GSTF7_ARATH*
accession n°: Q9SRY5
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 936.507 (0), 1030.521 (0), 1124.528 (0), 1143.599 (0),
1503.818 (0) }
[Click to access protein entry]
Spot: 2D-001KJ2 (arabidopsis)
pI: 5.92 Mw: 32050
%vol: 0.256991 %od: 0.235334
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.457 (0), 914.415 (0), 928.452 (0), 962.465 (0),
1039.459 (0), 1116.59 (0), 1187.678 (0), 1228.674 (0), 1249.694 (0),
1407.687 (0), 1447.768 (0), 1467.657 (0), 1475.777 (0), 1716.845 (0)
}
Spot: 2D-001KJ2 (arabidopsis)
pI: 5.92 Mw: 32050
%vol: 0.256991 %od: 0.235334
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.457 (0), 914.415 (0), 928.452 (0), 962.465 (0),
1039.459 (0), 1116.59 (0), 1187.678 (0), 1228.674 (0), 1249.694 (0),
1407.687 (0), 1447.768 (0), 1467.657 (0), 1475.777 (0), 1716.845 (0)
}
[Click to access protein entry]
Spot: 2D-001K5F (arabidopsis)
pI: 4.41 Mw: 55005
%vol: 0.12309 %od: 0.0843753
================
*CALR1_ARATH*
accession n°: O04151
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001K5F (arabidopsis)
pI: 4.41 Mw: 55005
%vol: 0.12309 %od: 0.0843753
================
*CALR1_ARATH*
accession n°: O04151
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KMW (arabidopsis)
pI: 5.82 Mw: 27447
%vol: 0.162557 %od: 0.121853
================
*GSTF6_ARATH*
accession n°: P42760
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1030.514 (0), 1124.492 (0), 1143.587 (0), 1475.765 (0),
1522.778 (0) }
Spot: 2D-001KMW (arabidopsis)
pI: 5.82 Mw: 27447
%vol: 0.162557 %od: 0.121853
================
*GSTF6_ARATH*
accession n°: P42760
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1030.514 (0), 1124.492 (0), 1143.587 (0), 1475.765 (0),
1522.778 (0) }
[Click to access protein entry]
Spot: 2D-001KBM (arabidopsis)
pI: 6.10 Mw: 43852
%vol: 0.298933 %od: 0.232866
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.442 (0), 914.367 (0), 928.432 (0), 1187.645 (0),
1228.645 (0), 1249.683 (0), 1261.62 (0), 1447.735 (0), 1465.773 (0),
1502.858 (0) }
Spot: 2D-001KBM (arabidopsis)
pI: 6.10 Mw: 43852
%vol: 0.298933 %od: 0.232866
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.442 (0), 914.367 (0), 928.432 (0), 1187.645 (0),
1228.645 (0), 1249.683 (0), 1261.62 (0), 1447.735 (0), 1465.773 (0),
1502.858 (0) }
[Click to access protein entry]
Spot: 2D-001K1O (arabidopsis)
pI: 5.26 Mw: 62957
%vol: 0.292681 %od: 1.21453
================
*BGL38_ARATH*
accession n°: P37702
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 866.532 (0), 1055.544 (0), 1077.568 (0), 1106.594 (0),
1314.585 (0), 1325.704 (0), 1331.66 (0), 1520.674 (0), 1744.796 (0),
1766.751 (0), 2152.907 (0) }
Spot: 2D-001K1O (arabidopsis)
pI: 5.26 Mw: 62957
%vol: 0.292681 %od: 1.21453
================
*BGL38_ARATH*
accession n°: P37702
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 866.532 (0), 1055.544 (0), 1077.568 (0), 1106.594 (0),
1314.585 (0), 1325.704 (0), 1331.66 (0), 1520.674 (0), 1744.796 (0),
1766.751 (0), 2152.907 (0) }
[Click to access protein entry]
Spot: 2D-001KKH (arabidopsis)
pI: 5.16 Mw: 30385
%vol: 0.0682532 %od: 0.063496
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KKH (arabidopsis)
pI: 5.16 Mw: 30385
%vol: 0.0682532 %od: 0.063496
================
*CAH2_ARATH*
accession n°: P42737
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KR5 (arabidopsis)
pI: 5.79 Mw: 15551
%vol: 0.12817 %od: 0.192109
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.473 (0), 935.506 (0), 1009.527 (0), 1137.633 (0),
1351.6 (0), 1548.861 (0) }
Spot: 2D-001KR5 (arabidopsis)
pI: 5.79 Mw: 15551
%vol: 0.12817 %od: 0.192109
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.473 (0), 935.506 (0), 1009.527 (0), 1137.633 (0),
1351.6 (0), 1548.861 (0) }
[Click to access protein entry]
Spot: 2D-001K1H (arabidopsis)
pI: 4.92 Mw: 64425
%vol: 0.150053 %od: 0.0934851
================
*HSP73_ARATH*
accession n°: O65719
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 1228.629 (0), 1231.64 (0), 1295.616 (0), 1308.662 (0),
1374.609 (0), 1431.736 (0), 1473.701 (0), 1522.822 (0), 1675.728 (0),
1680.835 (0), 2658.192 (0) }
Spot: 2D-001K1H (arabidopsis)
pI: 4.92 Mw: 64425
%vol: 0.150053 %od: 0.0934851
================
*HSP73_ARATH*
accession n°: O65719
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 1228.629 (0), 1231.64 (0), 1295.616 (0), 1308.662 (0),
1374.609 (0), 1431.736 (0), 1473.701 (0), 1522.822 (0), 1675.728 (0),
1680.835 (0), 2658.192 (0) }
[Click to access protein entry]
Spot: 2D-001KCA (arabidopsis)
pI: 6.83 Mw: 42694
%vol: 0.181314 %od: 0.109634
================
*GCST_ARATH*
accession n°: O65396
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1121.598 (0), 1277.685 (0), 1325.622 (0), 1407.68 (0),
1415.727 (0), 1419.749 (0), 1547.837 (0), 1622.81 (0), 1745.879 (0),
1815.004 (0), 1970.165 (0), 2450.09 (0) }
Spot: 2D-001KCA (arabidopsis)
pI: 6.83 Mw: 42694
%vol: 0.181314 %od: 0.109634
================
*GCST_ARATH*
accession n°: O65396
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1121.598 (0), 1277.685 (0), 1325.622 (0), 1407.68 (0),
1415.727 (0), 1419.749 (0), 1547.837 (0), 1622.81 (0), 1745.879 (0),
1815.004 (0), 1970.165 (0), 2450.09 (0) }
[Click to access protein entry]
Spot: 2D-001K18 (arabidopsis)
pI: 5.05 Mw: 67757
%vol: 0.236672 %od: 0.200561
================
*BIP2_ARATH*
accession n°: Q39043
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1155.523 (0), 1196.491 (0), 1296.455 (0), 1473.642 (0),
1536.662 (0), 1665.721 (0), 1861.829 (0), 1906.803 (0), 2691.022 (0)
}
================
*BIP1_ARATH*
accession n: Q9LKR3
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1155.523 (0), 1196.491 (0), 1296.455 (0), 1473.642 (0),
1536.662 (0), 1665.721 (0), 1861.829 (0), 1906.803 (0), 2052.821 (0),
2691.022 (0) }
Spot: 2D-001K18 (arabidopsis)
pI: 5.05 Mw: 67757
%vol: 0.236672 %od: 0.200561
================
*BIP2_ARATH*
accession n°: Q39043
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1155.523 (0), 1196.491 (0), 1296.455 (0), 1473.642 (0),
1536.662 (0), 1665.721 (0), 1861.829 (0), 1906.803 (0), 2691.022 (0)
}
================
*BIP1_ARATH*
accession n: Q9LKR3
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1155.523 (0), 1196.491 (0), 1296.455 (0), 1473.642 (0),
1536.662 (0), 1665.721 (0), 1861.829 (0), 1906.803 (0), 2052.821 (0),
2691.022 (0) }
[Click to access protein entry]
Spot: 2D-001KHU (arabidopsis)
pI: 6.02 Mw: 33897
%vol: 0.0522319 %od: 0.0235387
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.384 (0), 1021.498 (0), 1261.584 (0), 1407.677 (0),
1467.547 (0), 1475.747 (0), 1502.842 (0) }
Spot: 2D-001KHU (arabidopsis)
pI: 6.02 Mw: 33897
%vol: 0.0522319 %od: 0.0235387
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 910.384 (0), 1021.498 (0), 1261.584 (0), 1407.677 (0),
1467.547 (0), 1475.747 (0), 1502.842 (0) }
[Click to access protein entry]
Spot: 2D-001KIU (arabidopsis)
pI: 4.92 Mw: 32154
%vol: 0.30766 %od: 0.746412
================
*PSBO1_ARATH*
accession n°: P23321
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.495 (0), 950.558 (0), 1145.552 (0), 1203.65 (0),
1252.678 (0), 1362.646 (0), 1433.732 (0), 1450.734 (0), 1490.751 (0),
1562.796 (0), 1775.891 (0), 2019.001 (0), 2462.238 (0), 2604.356 (0)
}
================
*PSBO2_ARATH*
accession n: Q9S841
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.46 (0), 950.511 (0), 1252.568 (0), 1362.532 (0),
1433.597 (0), 1490.622 (0), 1562.685 (0), 2018.809 (0), 2531.073 (0),
2604.051 (0) }
Spot: 2D-001KIU (arabidopsis)
pI: 4.92 Mw: 32154
%vol: 0.30766 %od: 0.746412
================
*PSBO1_ARATH*
accession n°: P23321
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.495 (0), 950.558 (0), 1145.552 (0), 1203.65 (0),
1252.678 (0), 1362.646 (0), 1433.732 (0), 1450.734 (0), 1490.751 (0),
1562.796 (0), 1775.891 (0), 2019.001 (0), 2462.238 (0), 2604.356 (0)
}
================
*PSBO2_ARATH*
accession n: Q9S841
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 881.46 (0), 950.511 (0), 1252.568 (0), 1362.532 (0),
1433.597 (0), 1490.622 (0), 1562.685 (0), 2018.809 (0), 2531.073 (0),
2604.051 (0) }
[Click to access protein entry]
Spot: 2D-001KBF (arabidopsis)
pI: 5.26 Mw: 44124
%vol: 0.296198 %od: 0.294791
================
*GLNA2_ARATH*
accession n°: Q43127
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 879.411 (0), 899.538 (0), 937.445 (0), 948.4 (0), 1007.503 (0),
1027.624 (0), 1361.593 (0), 1397.663 (0), 1517.725 (0), 1581.818 (0),
1626.757 (0), 1763.917 (0), 1775.953 (0), 1817.858 (0), 2623.355 (0)
}
Spot: 2D-001KBF (arabidopsis)
pI: 5.26 Mw: 44124
%vol: 0.296198 %od: 0.294791
================
*GLNA2_ARATH*
accession n°: Q43127
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 879.411 (0), 899.538 (0), 937.445 (0), 948.4 (0), 1007.503 (0),
1027.624 (0), 1361.593 (0), 1397.663 (0), 1517.725 (0), 1581.818 (0),
1626.757 (0), 1763.917 (0), 1775.953 (0), 1817.858 (0), 2623.355 (0)
}
[Click to access protein entry]
Spot: 2D-001K32 (arabidopsis)
pI: 6.60 Mw: 58901
%vol: 0.375653 %od: 2.03006
================
*BGL18_ARATH*
accession n°: Q9SE50
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 951.181 (0), 1553.354 (0), 1788.497 (0), 1846.407 (0),
1894.423 (0), 1918.439 (0), 2383.443 (0), 2710.698 (0), 2884.279 (0)
}
Spot: 2D-001K32 (arabidopsis)
pI: 6.60 Mw: 58901
%vol: 0.375653 %od: 2.03006
================
*BGL18_ARATH*
accession n°: Q9SE50
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 951.181 (0), 1553.354 (0), 1788.497 (0), 1846.407 (0),
1894.423 (0), 1918.439 (0), 2383.443 (0), 2710.698 (0), 2884.279 (0)
}
[Click to access protein entry]
Spot: 2D-001KGE (arabidopsis)
pI: 5.25 Mw: 35689
%vol: 0.304013 %od: 0.570612
================
*ODPB_ARATH*
accession n°: Q38799
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1191.607 (0), 1266.745 (0), 1513.826 (0), 1811.882 (0)
}
Spot: 2D-001KGE (arabidopsis)
pI: 5.25 Mw: 35689
%vol: 0.304013 %od: 0.570612
================
*ODPB_ARATH*
accession n°: Q38799
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1191.607 (0), 1266.745 (0), 1513.826 (0), 1811.882 (0)
}
[Click to access protein entry]
Spot: 2D-001KL6 (arabidopsis)
pI: 5.79 Mw: 29705
%vol: 0.0730724 %od: 0.0506629
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.361 (0), 1170.621 (0), 1187.66 (0), 1228.652 (0),
1249.661 (0), 1475.753 (0) }
Spot: 2D-001KL6 (arabidopsis)
pI: 5.79 Mw: 29705
%vol: 0.0730724 %od: 0.0506629
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.361 (0), 1170.621 (0), 1187.66 (0), 1228.652 (0),
1249.661 (0), 1475.753 (0) }
[Click to access protein entry]
Spot: 2D-001K5T (arabidopsis)
pI: 5.12 Mw: 53441
%vol: 0.259857 %od: 0.480993
================
*ATPA_ARATH*
accession n°: P56757
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 876.447 (0), 901.446 (0), 903.539 (0), 1030.527 (0),
1101.548 (0), 1107.545 (0), 1209.625 (0), 1252.717 (0), 1416.79 (0),
1474.804 (0), 1553.752 (0), 1574.783 (0), 1942.062 (0)
}
Spot: 2D-001K5T (arabidopsis)
pI: 5.12 Mw: 53441
%vol: 0.259857 %od: 0.480993
================
*ATPA_ARATH*
accession n°: P56757
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 876.447 (0), 901.446 (0), 903.539 (0), 1030.527 (0),
1101.548 (0), 1107.545 (0), 1209.625 (0), 1252.717 (0), 1416.79 (0),
1474.804 (0), 1553.752 (0), 1574.783 (0), 1942.062 (0)
}
[Click to access protein entry]
Spot: 2D-001K1T (arabidopsis)
pI: 5.21 Mw: 63412
%vol: 0.113972 %od: 0.10967
================
*BGL38_ARATH*
accession n°: P37702
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1055.526 (0), 1077.53 (0), 1314.644 (0), 1331.675 (0),
1520.718 (0), 1744.866 (0), 1766.787 (0), 2153.031 (0)
}
Spot: 2D-001K1T (arabidopsis)
pI: 5.21 Mw: 63412
%vol: 0.113972 %od: 0.10967
================
*BGL38_ARATH*
accession n°: P37702
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1055.526 (0), 1077.53 (0), 1314.644 (0), 1331.675 (0),
1520.718 (0), 1744.866 (0), 1766.787 (0), 2153.031 (0)
}
[Click to access protein entry]
Spot: 2D-001KG0 (arabidopsis)
pI: 5.37 Mw: 36133
%vol: 0.326287 %od: 0.913153
================
*QORL_ARATH*
accession n°: Q9ZUC1
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1384.803 (0), 1487.867 (0), 1571.788 (0), 1723.874 (0),
2384.203 (0) }
Spot: 2D-001KG0 (arabidopsis)
pI: 5.37 Mw: 36133
%vol: 0.326287 %od: 0.913153
================
*QORL_ARATH*
accession n°: Q9ZUC1
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1384.803 (0), 1487.867 (0), 1571.788 (0), 1723.874 (0),
2384.203 (0) }
[Click to access protein entry]
Spot: 2D-001K6K (arabidopsis)
pI: 5.91 Mw: 52460
%vol: 0.29724 %od: 0.326756
================
*ATPAM_ARATH*
accession n°: P92549
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1235.474 (0), 1250.536 (0), 1438.694 (0), 1519.561 (0),
1537.665 (0), 1572.57 (0), 1738.756 (0), 2307.99 (0), 2367.11 (0)
}
Spot: 2D-001K6K (arabidopsis)
pI: 5.91 Mw: 52460
%vol: 0.29724 %od: 0.326756
================
*ATPAM_ARATH*
accession n°: P92549
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1235.474 (0), 1250.536 (0), 1438.694 (0), 1519.561 (0),
1537.665 (0), 1572.57 (0), 1738.756 (0), 2307.99 (0), 2367.11 (0)
}
[Click to access protein entry]
Spot: 2D-001KEU (arabidopsis)
pI: 5.90 Mw: 38674
%vol: 0.299715 %od: 0.340434
================
*MDHC1_ARATH*
accession n°: P93819
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KEU (arabidopsis)
pI: 5.90 Mw: 38674
%vol: 0.299715 %od: 0.340434
================
*MDHC1_ARATH*
accession n°: P93819
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KKM (arabidopsis)
pI: 5.28 Mw: 30336
%vol: 0.21036 %od: 0.174591
================
*Y2766_ARATH*
accession n°: O80934
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1121.51 (0), 1155.61 (0), 1186.549 (0), 1476.735 (0),
1585.806 (0), 1733.847 (0), 1809.961 (0), 1859.992 (0), 1938.072 (0)
}
Spot: 2D-001KKM (arabidopsis)
pI: 5.28 Mw: 30336
%vol: 0.21036 %od: 0.174591
================
*Y2766_ARATH*
accession n°: O80934
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1121.51 (0), 1155.61 (0), 1186.549 (0), 1476.735 (0),
1585.806 (0), 1733.847 (0), 1809.961 (0), 1859.992 (0), 1938.072 (0)
}
[Click to access protein entry]
Spot: 2D-001KMG (arabidopsis)
pI: 6.09 Mw: 28115
%vol: 0.284084 %od: 0.460041
================
*GSTF2_ARATH*
accession n°: P46422
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001KMG (arabidopsis)
pI: 6.09 Mw: 28115
%vol: 0.284084 %od: 0.460041
================
*GSTF2_ARATH*
accession n°: P46422
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001UMR (arabidopsis)
pI: 6.07 Mw: 17069
%vol: 0.396363 %od: 2.60604
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {Gm} {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.474 (0), 930.403 (0), 935.474 (0), 1009.501 (0),
1351.517 (0), 1548.718 (0), 2023.874 (0), 2069.945 (0), 2080.861 (0)
}
Spot: 2D-001UMR (arabidopsis)
pI: 6.07 Mw: 17069
%vol: 0.396363 %od: 2.60604
================
*RBS3B_ARATH*
accession n°: P99057
Identification Methods:
* MAPPING: {Gm} {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 907.474 (0), 930.403 (0), 935.474 (0), 1009.501 (0),
1351.517 (0), 1548.718 (0), 2023.874 (0), 2069.945 (0), 2080.861 (0)
}
[Click to access protein entry]
Spot: 2D-001K6S (arabidopsis)
pI: 4.33 Mw: 52029
%vol: 0.181314 %od: 0.185265
================
*CALR2_ARATH*
accession n°: Q38858
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001K6S (arabidopsis)
pI: 4.33 Mw: 52029
%vol: 0.181314 %od: 0.185265
================
*CALR2_ARATH*
accession n°: Q38858
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001KEG (arabidopsis)
pI: 6.12 Mw: 39236
%vol: 0.278353 %od: 0.252838
================
*PDI21_ARATH*
accession n°: O22263
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 947.403 (0), 1068.567 (0), 1090.559 (0), 1214.684 (0),
1221.604 (0), 1360.696 (0), 1708.788 (0) }
Spot: 2D-001KEG (arabidopsis)
pI: 6.12 Mw: 39236
%vol: 0.278353 %od: 0.252838
================
*PDI21_ARATH*
accession n°: O22263
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 947.403 (0), 1068.567 (0), 1090.559 (0), 1214.684 (0),
1221.604 (0), 1360.696 (0), 1708.788 (0) }
[Click to access protein entry]
Spot: 2D-001KOT (arabidopsis)
pI: 5.43 Mw: 21513
%vol: 0.296719 %od: 0.565436
================
*CP20C_ARATH*
accession n°: P34791
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 963.418 (0), 1142.494 (0), 1164.511 (0), 1201.554 (0),
1304.688 (0), 1359.676 (0), 1510.757 (0) }
Spot: 2D-001KOT (arabidopsis)
pI: 5.43 Mw: 21513
%vol: 0.296719 %od: 0.565436
================
*CP20C_ARATH*
accession n°: P34791
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 963.418 (0), 1142.494 (0), 1164.511 (0), 1201.554 (0),
1304.688 (0), 1359.676 (0), 1510.757 (0) }
[Click to access protein entry]
Spot: 2D-001KLF (arabidopsis)
pI: 5.79 Mw: 29370
%vol: 0.0857073 %od: 0.0608731
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.399 (0), 928.443 (0), 1116.592 (0), 1187.639 (0),
1228.664 (0), 1249.686 (0), 1447.77 (0) }
Spot: 2D-001KLF (arabidopsis)
pI: 5.79 Mw: 29370
%vol: 0.0857073 %od: 0.0608731
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.399 (0), 928.443 (0), 1116.592 (0), 1187.639 (0),
1228.664 (0), 1249.686 (0), 1447.77 (0) }
[Click to access protein entry]
Spot: 2D-001KBE (arabidopsis)
pI: 5.13 Mw: 43852
%vol: 0.321076 %od: 1.08452
================
*ACT8_ARATH*
accession n°: P99055
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 895.479 (0), 976.437 (0), 1008.572 (0), 1176.525 (0),
1182.504 (0), 1192.515 (0), 1198.691 (0), 1338.596 (0), 1475.663 (0),
1515.725 (0), 1547.736 (0), 1774.871 (0), 1855.888 (0), 1954.015 (0),
2199.024 (0) }
Spot: 2D-001KBE (arabidopsis)
pI: 5.13 Mw: 43852
%vol: 0.321076 %od: 1.08452
================
*ACT8_ARATH*
accession n°: P99055
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 895.479 (0), 976.437 (0), 1008.572 (0), 1176.525 (0),
1182.504 (0), 1192.515 (0), 1198.691 (0), 1338.596 (0), 1475.663 (0),
1515.725 (0), 1547.736 (0), 1774.871 (0), 1855.888 (0), 1954.015 (0),
2199.024 (0) }
[Click to access protein entry]
Spot: 2D-001KGO (arabidopsis)
pI: 5.30 Mw: 35469
%vol: 0.0660391 %od: 0.039628
================
*ARGI2_ARATH*
accession n°: Q9ZPF5
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 912.413 (0), 954.611 (0), 1100.674 (0), 1112.663 (0),
1174.525 (0), 1539.875 (0) }
Spot: 2D-001KGO (arabidopsis)
pI: 5.30 Mw: 35469
%vol: 0.0660391 %od: 0.039628
================
*ARGI2_ARATH*
accession n°: Q9ZPF5
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 912.413 (0), 954.611 (0), 1100.674 (0), 1112.663 (0),
1174.525 (0), 1539.875 (0) }
[Click to access protein entry]
Spot: 2D-001KAA (arabidopsis)
pI: 5.22 Mw: 46361
%vol: 0.210751 %od: 0.116691
================
*MB31_ARATH*
accession n°: O04309
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 882.479 (0), 906.482 (0), 1001.455 (0), 1359.772 (0),
1469.724 (0), 1567.621 (0), 1597.804 (0), 1607.773 (0), 1636.742 (0),
1839.87 (0), 1864.816 (0) }
Spot: 2D-001KAA (arabidopsis)
pI: 5.22 Mw: 46361
%vol: 0.210751 %od: 0.116691
================
*MB31_ARATH*
accession n°: O04309
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 882.479 (0), 906.482 (0), 1001.455 (0), 1359.772 (0),
1469.724 (0), 1567.621 (0), 1597.804 (0), 1607.773 (0), 1636.742 (0),
1839.87 (0), 1864.816 (0) }
[Click to access protein entry]
Spot: 2D-001KNM (arabidopsis)
pI: 5.24 Mw: 25036
%vol: 0.208797 %od: 0.327409
================
*PSBP1_ARATH*
accession n°: Q42029
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1203.533 (0), 1351.6 (0), 1588.579 (0), 2068.886 (0),
2210.994 (0), 2360.058 (0), 2402 (0) }
Spot: 2D-001KNM (arabidopsis)
pI: 5.24 Mw: 25036
%vol: 0.208797 %od: 0.327409
================
*PSBP1_ARATH*
accession n°: Q42029
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1203.533 (0), 1351.6 (0), 1588.579 (0), 2068.886 (0),
2210.994 (0), 2360.058 (0), 2402 (0) }
[Click to access protein entry]
Spot: 2D-001KHE (arabidopsis)
pI: 5.95 Mw: 34390
%vol: 0.261029 %od: 0.186658
================
*MDHM1_ARATH*
accession n°: Q9ZP06
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1219.561 (0), 1347.653 (0), 1590.823 (0), 3007.042 (0)
}
Spot: 2D-001KHE (arabidopsis)
pI: 5.95 Mw: 34390
%vol: 0.261029 %od: 0.186658
================
*MDHM1_ARATH*
accession n°: Q9ZP06
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1219.561 (0), 1347.653 (0), 1590.823 (0), 3007.042 (0)
}
[Click to access protein entry]
Spot: 2D-001K9K (arabidopsis)
pI: 5.01 Mw: 47034
%vol: 0.318992 %od: 0.830877
================
*RCA_ARATH*
accession n°: P10896
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 895.385 (0), 1007.495 (0), 1023.491 (0), 1033.479 (0),
1059.563 (0), 1135.576 (0), 1179.6 (0), 1228.556 (0), 1697.786 (0),
2347.234 (0) }
Spot: 2D-001K9K (arabidopsis)
pI: 5.01 Mw: 47034
%vol: 0.318992 %od: 0.830877
================
*RCA_ARATH*
accession n°: P10896
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 895.385 (0), 1007.495 (0), 1023.491 (0), 1033.479 (0),
1059.563 (0), 1135.576 (0), 1179.6 (0), 1228.556 (0), 1697.786 (0),
2347.234 (0) }
[Click to access protein entry]
Spot: 2D-001KKV (arabidopsis)
pI: 6.47 Mw: 29849
%vol: 0.324724 %od: 0.84319
================
*VSP2_ARATH*
accession n°: O82122
Identification Methods:
* MAPPING: {PMF}
Spot: 2D-001KKV (arabidopsis)
pI: 6.47 Mw: 29849
%vol: 0.324724 %od: 0.84319
================
*VSP2_ARATH*
accession n°: O82122
Identification Methods:
* MAPPING: {PMF}
[Click to access protein entry]
Spot: 2D-001KMV (arabidopsis)
pI: 5.25 Mw: 27447
%vol: 0.309093 %od: 0.576443
================
*CH10C_ARATH*
accession n°: O65282
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 970.506 (0), 1008.49 (0), 1149.616 (0), 1193.62 (0),
1204.669 (0), 1371.648 (0), 1618.91 (0), 1662.934 (0), 1861.081 (0),
2083.142 (0), 2193.292 (0), 2540.399 (0), 3727.132 (0)
}
Spot: 2D-001KMV (arabidopsis)
pI: 5.25 Mw: 27447
%vol: 0.309093 %od: 0.576443
================
*CH10C_ARATH*
accession n°: O65282
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 970.506 (0), 1008.49 (0), 1149.616 (0), 1193.62 (0),
1204.669 (0), 1371.648 (0), 1618.91 (0), 1662.934 (0), 1861.081 (0),
2083.142 (0), 2193.292 (0), 2540.399 (0), 3727.132 (0)
}
[Click to access protein entry]
Spot: 2D-001KHD (arabidopsis)
pI: 6.00 Mw: 34532
%vol: 0.226512 %od: 0.136907
================
*CYSKP_ARATH*
accession n°: P47999
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1086.595 (0), 1334.751 (0), 1345.735 (0), 1355.725 (0),
1493.762 (0), 1846.853 (0) }
Spot: 2D-001KHD (arabidopsis)
pI: 6.00 Mw: 34532
%vol: 0.226512 %od: 0.136907
================
*CYSKP_ARATH*
accession n°: P47999
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1086.595 (0), 1334.751 (0), 1345.735 (0), 1355.725 (0),
1493.762 (0), 1846.853 (0) }
[Click to access protein entry]
Spot: 2D-001KB5 (arabidopsis)
pI: 5.44 Mw: 44398
%vol: 0.125695 %od: 0.107661
================
*EFTU_ARATH*
accession n°: P17745
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.564 (0), 1157.672 (0), 1307.648 (0), 1405.661 (0),
1435.686 (0), 1681.957 (0), 1810.89 (0), 1919.894 (0), 2066.944 (0)
}
Spot: 2D-001KB5 (arabidopsis)
pI: 5.44 Mw: 44398
%vol: 0.125695 %od: 0.107661
================
*EFTU_ARATH*
accession n°: P17745
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.564 (0), 1157.672 (0), 1307.648 (0), 1405.661 (0),
1435.686 (0), 1681.957 (0), 1810.89 (0), 1919.894 (0), 2066.944 (0)
}
[Click to access protein entry]
Spot: 2D-001KCZ (arabidopsis)
pI: 6.33 Mw: 41395
%vol: 0.239277 %od: 0.176757
================
*CHLI_ARATH*
accession n°: P16127
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1066.542 (0), 1102.537 (0), 1202.532 (0), 1218.576 (0),
1261.619 (0), 1559.744 (0), 1632.815 (0), 1669.844 (0), 2353.23 (0)
}
Spot: 2D-001KCZ (arabidopsis)
pI: 6.33 Mw: 41395
%vol: 0.239277 %od: 0.176757
================
*CHLI_ARATH*
accession n°: P16127
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1066.542 (0), 1102.537 (0), 1202.532 (0), 1218.576 (0),
1261.619 (0), 1559.744 (0), 1632.815 (0), 1669.844 (0), 2353.23 (0)
}
[Click to access protein entry]
Spot: 2D-001K74 (arabidopsis)
pI: 5.64 Mw: 51074
%vol: 0.324333 %od: 1.91242
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.433 (0), 928.484 (0), 962.497 (0), 1021.564 (0),
1039.502 (0), 1116.616 (0), 1187.697 (0), 1228.692 (0), 1249.714 (0),
1261.654 (0), 1324.715 (0), 1447.831 (0), 1502.886 (0), 1546.757 (0),
1810.846 (0), 1819.933 (0), 1866.902 (0), 1966.893 (0), 1999.002 (0),
2185.971 (0) }
Spot: 2D-001K74 (arabidopsis)
pI: 5.64 Mw: 51074
%vol: 0.324333 %od: 1.91242
================
*RBL_ARATH*
accession n°: O03042
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 914.433 (0), 928.484 (0), 962.497 (0), 1021.564 (0),
1039.502 (0), 1116.616 (0), 1187.697 (0), 1228.692 (0), 1249.714 (0),
1261.654 (0), 1324.715 (0), 1447.831 (0), 1502.886 (0), 1546.757 (0),
1810.846 (0), 1819.933 (0), 1866.902 (0), 1966.893 (0), 1999.002 (0),
2185.971 (0) }
[Click to access protein entry]
Spot: 2D-001K1E (arabidopsis)
pI: 4.97 Mw: 65407
%vol: 0.227293 %od: 0.16429
================
*HSP71_ARATH*
accession n°: P22953
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 1228.649 (0), 1294.617 (0), 1426.791 (0), 1473.701 (0),
1659.845 (0), 1663.782 (0), 1675.735 (0), 1680.826 (0), 1838.871 (0),
2658.184 (0) }
Spot: 2D-001K1E (arabidopsis)
pI: 4.97 Mw: 65407
%vol: 0.227293 %od: 0.16429
================
*HSP71_ARATH*
accession n°: P22953
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 1228.649 (0), 1294.617 (0), 1426.791 (0), 1473.701 (0),
1659.845 (0), 1663.782 (0), 1675.735 (0), 1680.826 (0), 1838.871 (0),
2658.184 (0) }
[Click to access protein entry]
Spot: 2D-001KDP (arabidopsis)
pI: 5.17 Mw: 40135
%vol: 0.259336 %od: 0.429056
================
*G3PA_ARATH*
accession n°: P25856
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 928.541 (0), 1037.609 (0), 1254.62 (0), 1382.73 (0),
1384.814 (0), 1571.741 (0), 1627.913 (0), 1755.955 (0), 1786.837 (0),
1833.984 (0), 2585.278 (0) }
Spot: 2D-001KDP (arabidopsis)
pI: 5.17 Mw: 40135
%vol: 0.259336 %od: 0.429056
================
*G3PA_ARATH*
accession n°: P25856
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 928.541 (0), 1037.609 (0), 1254.62 (0), 1382.73 (0),
1384.814 (0), 1571.741 (0), 1627.913 (0), 1755.955 (0), 1786.837 (0),
1833.984 (0), 2585.278 (0) }
[Click to access protein entry]
Spot: 2D-001KPS (arabidopsis)
pI: 8.07 Mw: 27149
%vol: 0.0646059 %od: 0.0466047
================
*PSBQ1_ARATH*
accession n°: Q9XFT3
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1261.641 (0), 1295.779 (0), 1378.75 (0), 1389.711 (0),
1498.745 (0), 1573.862 (0), 1670.856 (0), 1844.964 (0), 1989.978 (0)
}
Spot: 2D-001KPS (arabidopsis)
pI: 8.07 Mw: 27149
%vol: 0.0646059 %od: 0.0466047
================
*PSBQ1_ARATH*
accession n°: Q9XFT3
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1261.641 (0), 1295.779 (0), 1378.75 (0), 1389.711 (0),
1498.745 (0), 1573.862 (0), 1670.856 (0), 1844.964 (0), 1989.978 (0)
}
[Click to access protein entry]
Spot: 2D-001KML (arabidopsis)
pI: 5.49 Mw: 27934
%vol: 0.151746 %od: 0.1593
================
*GSTFA_ARATH*
accession n°: P42761
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1118.598 (0), 1390.755 (0), 1409.816 (0), 1454.721 (0),
1577.838 (0), 1924.917 (0) }
Spot: 2D-001KML (arabidopsis)
pI: 5.49 Mw: 27934
%vol: 0.151746 %od: 0.1593
================
*GSTFA_ARATH*
accession n°: P42761
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1118.598 (0), 1390.755 (0), 1409.816 (0), 1454.721 (0),
1577.838 (0), 1924.917 (0) }
[Click to access protein entry]
Spot: 2D-001KDQ (arabidopsis)
pI: 6.51 Mw: 40135
%vol: 0.347257 %od: 0.571326
================
*KPPR_ARATH*
accession n°: P25697
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 870.415 (0), 1166.595 (0), 1272.735 (0), 1359.648 (0),
1420.711 (0), 1428.837 (0), 1475.805 (0), 1488.671 (0), 1668.928 (0),
1684.892 (0), 1689.868 (0), 1961.071 (0) }
Spot: 2D-001KDQ (arabidopsis)
pI: 6.51 Mw: 40135
%vol: 0.347257 %od: 0.571326
================
*KPPR_ARATH*
accession n°: P25697
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 870.415 (0), 1166.595 (0), 1272.735 (0), 1359.648 (0),
1420.711 (0), 1428.837 (0), 1475.805 (0), 1488.671 (0), 1668.928 (0),
1684.892 (0), 1689.868 (0), 1961.071 (0) }
SWISS-2DPAGE Viewer
|
Back to the |
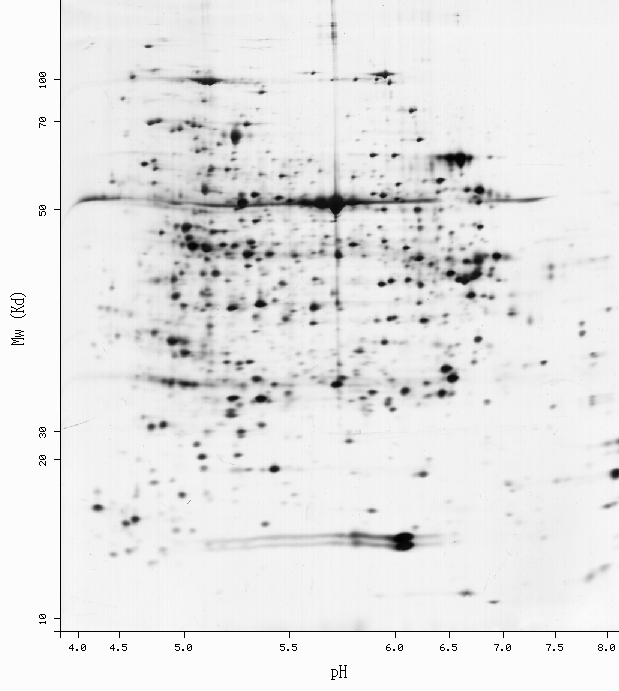
-Get more information by dragging your mouse pointer over any highlighted spot -Click on a highlighted spot to access all its associated protein entries
Back to the