Attention: World-2DPAGE is no longer maintained.
[Click to access protein entry]
Spot: 2D-001DUU (dld1_human)
pI: 5.49 Mw: 53373
%vol: 0.0889513 %od: 0.103578
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 877.468 (0), 995.567 (0), 1188.586 (0), 1191.613 (0),
1236.537 (0), 1359.691 (0), 1370.719 (0), 1639.774 (0)
}
Spot: 2D-001DUU (dld1_human)
pI: 5.49 Mw: 53373
%vol: 0.0889513 %od: 0.103578
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 877.468 (0), 995.567 (0), 1188.586 (0), 1191.613 (0),
1236.537 (0), 1359.691 (0), 1370.719 (0), 1639.774 (0)
}
[Click to access protein entry]
Spot: 2D-001EKT (dld1_human)
pI: 5.60 Mw: 29890
%vol: 0.0777495 %od: 0.113735
================
*ANXA4_HUMAN*
accession n°: P09525
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1134.49 (0), 1371.65 (0), 1379.67 (0), 1597.77 (0), 1692.89 (0)
}
Spot: 2D-001EKT (dld1_human)
pI: 5.60 Mw: 29890
%vol: 0.0777495 %od: 0.113735
================
*ANXA4_HUMAN*
accession n°: P09525
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1134.49 (0), 1371.65 (0), 1379.67 (0), 1597.77 (0), 1692.89 (0)
}
[Click to access protein entry]
Spot: 2D-001DK7 (dld1_human)
pI: 5.17 Mw: 69894
%vol: 0.0484921 %od: 0.0284859
================
*HSP7C_HUMAN*
accession n°: P11142
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1253.53 (0), 1481.7 (0), 1487.59 (0), 1981.83 (0)
}
Spot: 2D-001DK7 (dld1_human)
pI: 5.17 Mw: 69894
%vol: 0.0484921 %od: 0.0284859
================
*HSP7C_HUMAN*
accession n°: P11142
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1253.53 (0), 1481.7 (0), 1487.59 (0), 1981.83 (0)
}
[Click to access protein entry]
Spot: 2D-001DJI (dld1_human)
pI: 5.27 Mw: 71362
%vol: 0.0680584 %od: 0.108766
================
*GRP75_HUMAN*
accession n°: P38646
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 958.08 (0), 1148.077 (0), 1242.261 (0), 1290.268 (0),
1361.326 (0), 1450.365 (0), 1462.352 (0), 1592.513 (0), 1694.443 (0),
2055.579 (0) }
Spot: 2D-001DJI (dld1_human)
pI: 5.27 Mw: 71362
%vol: 0.0680584 %od: 0.108766
================
*GRP75_HUMAN*
accession n°: P38646
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 958.08 (0), 1148.077 (0), 1242.261 (0), 1290.268 (0),
1361.326 (0), 1450.365 (0), 1462.352 (0), 1592.513 (0), 1694.443 (0),
2055.579 (0) }
[Click to access protein entry]
Spot: 2D-001ERN (dld1_human)
pI: 5.55 Mw: 25144
%vol: 0.0581094 %od: 0.0709407
================
*SPTB1_HUMAN*
accession n°: P11277
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001ERN (dld1_human)
pI: 5.55 Mw: 25144
%vol: 0.0581094 %od: 0.0709407
================
*SPTB1_HUMAN*
accession n°: P11277
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DKR (dld1_human)
pI: 5.62 Mw: 67048
%vol: 0.0764229 %od: 0.211475
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DKR (dld1_human)
pI: 5.62 Mw: 67048
%vol: 0.0764229 %od: 0.211475
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EXB (dld1_human)
pI: 4.87 Mw: 21107
%vol: 0.0130442 %od: 0.00678338
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EXB (dld1_human)
pI: 4.87 Mw: 21107
%vol: 0.0130442 %od: 0.00678338
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EWB (dld1_human)
pI: 5.38 Mw: 21646
%vol: 0.09411 %od: 0.372523
================
*PRDX2_HUMAN*
accession n°: P32119
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 972.658 (0), 1179.765 (0), 1211.806 (0), 1334.906 (0),
1929.196 (0) }
Spot: 2D-001EWB (dld1_human)
pI: 5.38 Mw: 21646
%vol: 0.09411 %od: 0.372523
================
*PRDX2_HUMAN*
accession n°: P32119
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 972.658 (0), 1179.765 (0), 1211.806 (0), 1334.906 (0),
1929.196 (0) }
[Click to access protein entry]
Spot: 2D-001CZK (dld1_human)
pI: 5.89 Mw: 129569
%vol: 0.0605046 %od: 0.0998582
================
*VINC_HUMAN*
accession n°: P18206
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 923.447 (0), 944.47 (0), 1170.638 (0), 1476.826 (0),
1484.808 (0), 2036.064 (0), 2076.148 (0), 2447.124 (0), 3023.369 (0)
}
Spot: 2D-001CZK (dld1_human)
pI: 5.89 Mw: 129569
%vol: 0.0605046 %od: 0.0998582
================
*VINC_HUMAN*
accession n°: P18206
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 923.447 (0), 944.47 (0), 1170.638 (0), 1476.826 (0),
1484.808 (0), 2036.064 (0), 2076.148 (0), 2447.124 (0), 3023.369 (0)
}
[Click to access protein entry]
Spot: 2D-001ERO (dld1_human)
pI: 5.59 Mw: 25086
%vol: 0.0435176 %od: 0.0642969
================
*PGDH_HUMAN*
accession n°: P15428
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001ERO (dld1_human)
pI: 5.59 Mw: 25086
%vol: 0.0435176 %od: 0.0642969
================
*PGDH_HUMAN*
accession n°: P15428
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E1B (dld1_human)
pI: 6.60 Mw: 46694
%vol: 0.0465391 %od: 0.025203
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001E1B (dld1_human)
pI: 6.60 Mw: 46694
%vol: 0.0465391 %od: 0.025203
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DWU (dld1_human)
pI: 6.65 Mw: 51091
%vol: 0.0795919 %od: 0.135812
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DWU (dld1_human)
pI: 6.65 Mw: 51091
%vol: 0.0795919 %od: 0.135812
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DL5 (dld1_human)
pI: 5.69 Mw: 66700
%vol: 0.0774178 %od: 0.192045
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DL5 (dld1_human)
pI: 5.69 Mw: 66700
%vol: 0.0774178 %od: 0.192045
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DV9 (dld1_human)
pI: 5.34 Mw: 53236
%vol: 0.0363321 %od: 0.0157283
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DV9 (dld1_human)
pI: 5.34 Mw: 53236
%vol: 0.0363321 %od: 0.0157283
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DYA (dld1_human)
pI: 6.90 Mw: 50309
%vol: 0.0363691 %od: 0.00558582
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DYA (dld1_human)
pI: 6.90 Mw: 50309
%vol: 0.0363691 %od: 0.00558582
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DU1 (dld1_human)
pI: 4.84 Mw: 54064
%vol: 0.0627523 %od: 0.0462217
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
Spot: 2D-001DU1 (dld1_human)
pI: 4.84 Mw: 54064
%vol: 0.0627523 %od: 0.0462217
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
[Click to access protein entry]
Spot: 2D-001EUM (dld1_human)
pI: 5.42 Mw: 22876
%vol: 0.0973526 %od: 0.373917
================
*GSTP1_HUMAN*
accession n°: P09211
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1337.63 (0), 1534.71 (0), 1883.83 (0), 2126.03 (0), 2132.85 (0),
2154.92 (0) }
Spot: 2D-001EUM (dld1_human)
pI: 5.42 Mw: 22876
%vol: 0.0973526 %od: 0.373917
================
*GSTP1_HUMAN*
accession n°: P09211
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1337.63 (0), 1534.71 (0), 1883.83 (0), 2126.03 (0), 2132.85 (0),
2154.92 (0) }
[Click to access protein entry]
Spot: 2D-001EUB (dld1_human)
pI: 6.31 Mw: 23409
%vol: 0.00541667 %od: 0.00106638
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EUB (dld1_human)
pI: 6.31 Mw: 23409
%vol: 0.00541667 %od: 0.00106638
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001E8K (dld1_human)
pI: 4.94 Mw: 38893
%vol: 0.0266044 %od: 0.0256973
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.391 (0), 1041.58 (0), 1122.58 (0), 1222.68 (0), 1554.77 (0),
1674.76 (0) }
Spot: 2D-001E8K (dld1_human)
pI: 4.94 Mw: 38893
%vol: 0.0266044 %od: 0.0256973
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.391 (0), 1041.58 (0), 1122.58 (0), 1222.68 (0), 1554.77 (0),
1674.76 (0) }
[Click to access protein entry]
Spot: 2D-001DUP (dld1_human)
pI: 5.60 Mw: 53100
%vol: 0.0969473 %od: 0.418882
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 877.13 (0), 995.204 (0), 1188.162 (0), 1191.238 (0),
1236.165 (0), 1341.334 (0), 1359.316 (0), 1370.345 (0), 1639.422 (0),
1680.424 (0) }
Spot: 2D-001DUP (dld1_human)
pI: 5.60 Mw: 53100
%vol: 0.0969473 %od: 0.418882
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 877.13 (0), 995.204 (0), 1188.162 (0), 1191.238 (0),
1236.165 (0), 1341.334 (0), 1359.316 (0), 1370.345 (0), 1639.422 (0),
1680.424 (0) }
[Click to access protein entry]
Spot: 2D-001DX7 (dld1_human)
pI: 6.54 Mw: 51091
%vol: 0.0590306 %od: 0.0782531
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DX7 (dld1_human)
pI: 6.54 Mw: 51091
%vol: 0.0590306 %od: 0.0782531
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EKL (dld1_human)
pI: 6.11 Mw: 30167
%vol: 0.0310262 %od: 0.0176317
================
*CEL3B_HUMAN*
accession n°: P08861
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1079.58 (0), 1095.56 (0), 1871.98 (0) }
Spot: 2D-001EKL (dld1_human)
pI: 6.11 Mw: 30167
%vol: 0.0310262 %od: 0.0176317
================
*CEL3B_HUMAN*
accession n°: P08861
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1079.58 (0), 1095.56 (0), 1871.98 (0) }
[Click to access protein entry]
Spot: 2D-001F1V (dld1_human)
pI: 5.64 Mw: 16489
%vol: 0.0620153 %od: 0.229617
================
*SODC_HUMAN*
accession n°: P00441
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.9 (0), 1225.32 (0), 1501.47 (0), 1502.19 (0), 2515.07 (0)
}
Spot: 2D-001F1V (dld1_human)
pI: 5.64 Mw: 16489
%vol: 0.0620153 %od: 0.229617
================
*SODC_HUMAN*
accession n°: P00441
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.9 (0), 1225.32 (0), 1501.47 (0), 1502.19 (0), 2515.07 (0)
}
[Click to access protein entry]
Spot: 2D-001DRK (dld1_human)
pI: 5.07 Mw: 56917
%vol: 0.0548668 %od: 0.036896
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DRK (dld1_human)
pI: 5.07 Mw: 56917
%vol: 0.0548668 %od: 0.036896
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E15 (dld1_human)
pI: 6.90 Mw: 46815
%vol: 0.0315789 %od: 0.0224804
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001E15 (dld1_human)
pI: 6.90 Mw: 46815
%vol: 0.0315789 %od: 0.0224804
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001E6P (dld1_human)
pI: 5.04 Mw: 41700
%vol: 0.0676899 %od: 0.0923318
================
*K2C8_HUMAN*
accession n°: P05787
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 932.471 (0), 1079.52 (0), 1129.61 (0), 1344.67 (0), 1352.7 (0),
1419.73 (0), 1762.99 (0), 1879.85 (0), 2124.99 (0)
}
Spot: 2D-001E6P (dld1_human)
pI: 5.04 Mw: 41700
%vol: 0.0676899 %od: 0.0923318
================
*K2C8_HUMAN*
accession n°: P05787
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 932.471 (0), 1079.52 (0), 1129.61 (0), 1344.67 (0), 1352.7 (0),
1419.73 (0), 1762.99 (0), 1879.85 (0), 2124.99 (0)
}
[Click to access protein entry]
Spot: 2D-001F5Y (dld1_human)
pI: 4.50 Mw: 13360
%vol: 0.0190136 %od: 0.0285198
================
*ATPD_HUMAN*
accession n°: P30049
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F5Y (dld1_human)
pI: 4.50 Mw: 13360
%vol: 0.0190136 %od: 0.0285198
================
*ATPD_HUMAN*
accession n°: P30049
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E4E (dld1_human)
pI: 5.11 Mw: 43451
%vol: 0.0685006 %od: 0.107388
================
*K2C8_HUMAN*
accession n°: P05787
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1079.51 (0), 1129.59 (0), 1344.65 (0), 1419.72 (0)
}
Spot: 2D-001E4E (dld1_human)
pI: 5.11 Mw: 43451
%vol: 0.0685006 %od: 0.107388
================
*K2C8_HUMAN*
accession n°: P05787
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1079.51 (0), 1129.59 (0), 1344.65 (0), 1419.72 (0)
}
[Click to access protein entry]
Spot: 2D-001EPH (dld1_human)
pI: 5.65 Mw: 26330
%vol: 0.0830924 %od: 0.165376
================
*ERP29_HUMAN*
accession n°: P30040
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EPH (dld1_human)
pI: 5.65 Mw: 26330
%vol: 0.0830924 %od: 0.165376
================
*ERP29_HUMAN*
accession n°: P30040
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EU0 (dld1_human)
pI: 7.55 Mw: 23087
%vol: 0.103543 %od: 0.389773
================
*PRDX1_HUMAN*
accession n°: Q06830
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.418 (0), 920.519 (0), 980.52 (0), 1107.59 (0), 1180.56 (0),
1196.61 (0), 1211.66 (0), 1638.83 (0) }
Spot: 2D-001EU0 (dld1_human)
pI: 7.55 Mw: 23087
%vol: 0.103543 %od: 0.389773
================
*PRDX1_HUMAN*
accession n°: Q06830
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.418 (0), 920.519 (0), 980.52 (0), 1107.59 (0), 1180.56 (0),
1196.61 (0), 1211.66 (0), 1638.83 (0) }
[Click to access protein entry]
Spot: 2D-001EWL (dld1_human)
pI: 4.79 Mw: 21586
%vol: 0.00549036 %od: 0.00257709
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EWL (dld1_human)
pI: 4.79 Mw: 21586
%vol: 0.00549036 %od: 0.00257709
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001E8S (dld1_human)
pI: 5.00 Mw: 38444
%vol: 0.0387642 %od: 0.080384
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.487 (0), 1029.628 (0), 1041.634 (0), 1073.614 (0),
1122.597 (0), 1222.662 (0), 1227.617 (0), 1370.614 (0), 1389.699 (0),
1554.759 (0), 1674.776 (0), 1833.946 (0), 1904.903 (0), 2794.351 (0)
}
Spot: 2D-001E8S (dld1_human)
pI: 5.00 Mw: 38444
%vol: 0.0387642 %od: 0.080384
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.487 (0), 1029.628 (0), 1041.634 (0), 1073.614 (0),
1122.597 (0), 1222.662 (0), 1227.617 (0), 1370.614 (0), 1389.699 (0),
1554.759 (0), 1674.776 (0), 1833.946 (0), 1904.903 (0), 2794.351 (0)
}
[Click to access protein entry]
Spot: 2D-001DZF (dld1_human)
pI: 4.99 Mw: 48281
%vol: 0.0944417 %od: 0.169233
================
*ATPB_HUMAN*
accession n°: P06576
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1038.766 (0), 1406.92 (0), 1436.003 (0), 1440.018 (0),
1618.113 (0), 1651.2 (0), 1922.308 (0), 1988.368 (0)
}
Spot: 2D-001DZF (dld1_human)
pI: 4.99 Mw: 48281
%vol: 0.0944417 %od: 0.169233
================
*ATPB_HUMAN*
accession n°: P06576
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1038.766 (0), 1406.92 (0), 1436.003 (0), 1440.018 (0),
1618.113 (0), 1651.2 (0), 1922.308 (0), 1988.368 (0)
}
[Click to access protein entry]
Spot: 2D-001E2N (dld1_human)
pI: 6.85 Mw: 45160
%vol: 0.0857823 %od: 0.143714
================
*FUMH_HUMAN*
accession n°: P07954
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 957.477 (0), 1214.67 (0), 1498.97 (0), 1763.93 (0)
}
Spot: 2D-001E2N (dld1_human)
pI: 6.85 Mw: 45160
%vol: 0.0857823 %od: 0.143714
================
*FUMH_HUMAN*
accession n°: P07954
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 957.477 (0), 1214.67 (0), 1498.97 (0), 1763.93 (0)
}
[Click to access protein entry]
Spot: 2D-001EEW (dld1_human)
pI: 5.57 Mw: 34211
%vol: 0.0355584 %od: 0.0150631
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EEW (dld1_human)
pI: 5.57 Mw: 34211
%vol: 0.0355584 %od: 0.0150631
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EBA (dld1_human)
pI: 6.32 Mw: 36667
%vol: 0.0967631 %od: 0.122813
================
*AK1A1_HUMAN*
accession n°: P14550
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 897.547 (0), 928.507 (0), 1375.75 (0), 1551.76 (0)
}
Spot: 2D-001EBA (dld1_human)
pI: 6.32 Mw: 36667
%vol: 0.0967631 %od: 0.122813
================
*AK1A1_HUMAN*
accession n°: P14550
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 897.547 (0), 928.507 (0), 1375.75 (0), 1551.76 (0)
}
[Click to access protein entry]
Spot: 2D-001ER3 (dld1_human)
pI: 6.64 Mw: 25260
%vol: 0.106491 %od: 0.532087
================
*TPIS_HUMAN*
accession n°: P60174
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1414.79 (0), 1458.76 (0), 1466.72 (0), 2192.04 (0)
}
Spot: 2D-001ER3 (dld1_human)
pI: 6.64 Mw: 25260
%vol: 0.106491 %od: 0.532087
================
*TPIS_HUMAN*
accession n°: P60174
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1414.79 (0), 1458.76 (0), 1466.72 (0), 2192.04 (0)
}
[Click to access protein entry]
Spot: 2D-001E5J (dld1_human)
pI: 5.15 Mw: 41700
%vol: 0.0920834 %od: 0.303067
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.504 (0), 1132.57 (0), 1198.7 (0), 1516.72 (0), 1790.89 (0),
1954.05 (0), 2231.02 (0) }
Spot: 2D-001E5J (dld1_human)
pI: 5.15 Mw: 41700
%vol: 0.0920834 %od: 0.303067
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.504 (0), 1132.57 (0), 1198.7 (0), 1516.72 (0), 1790.89 (0),
1954.05 (0), 2231.02 (0) }
[Click to access protein entry]
Spot: 2D-001F9A (dld1_human)
pI: 4.92 Mw: 10391
%vol: 0.0651843 %od: 0.183556
================
*THIO_HUMAN*
accession n°: P10599
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F9A (dld1_human)
pI: 4.92 Mw: 10391
%vol: 0.0651843 %od: 0.183556
================
*THIO_HUMAN*
accession n°: P10599
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001FB0 (dld1_human)
pI: 5.73 Mw: 9254
%vol: 0.0402381 %od: 0.0187091
================
*TRFE_HUMAN*
accession n°: P02787
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001FB0 (dld1_human)
pI: 5.73 Mw: 9254
%vol: 0.0402381 %od: 0.0187091
================
*TRFE_HUMAN*
accession n°: P02787
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E9S (dld1_human)
pI: 6.32 Mw: 37524
%vol: 0.0718538 %od: 0.07578
================
*ACADS_HUMAN*
accession n°: P16219
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001E9S (dld1_human)
pI: 6.32 Mw: 37524
%vol: 0.0718538 %od: 0.07578
================
*ACADS_HUMAN*
accession n°: P16219
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DDN (dld1_human)
pI: 6.03 Mw: 86045
%vol: 0.0677268 %od: 0.176427
================
*EZRI_HUMAN*
accession n°: P15311
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.525 (0), 987.508 (0), 1002.5 (0), 1104.55 (0), 1120.55 (0),
1182.57 (0), 1310.69 (0), 1651.83 (0) }
Spot: 2D-001DDN (dld1_human)
pI: 6.03 Mw: 86045
%vol: 0.0677268 %od: 0.176427
================
*EZRI_HUMAN*
accession n°: P15311
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.525 (0), 987.508 (0), 1002.5 (0), 1104.55 (0), 1120.55 (0),
1182.57 (0), 1310.69 (0), 1651.83 (0) }
[Click to access protein entry]
Spot: 2D-001EDR (dld1_human)
pI: 4.82 Mw: 34718
%vol: 0.053356 %od: 0.0969426
================
*NPM_HUMAN*
accession n°: P06748
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1568.71 (0), 1583.85 (0), 2160.98 (0), 2227.15 (0), 2243.2 (0),
2259.16 (0) }
Spot: 2D-001EDR (dld1_human)
pI: 4.82 Mw: 34718
%vol: 0.053356 %od: 0.0969426
================
*NPM_HUMAN*
accession n°: P06748
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1568.71 (0), 1583.85 (0), 2160.98 (0), 2227.15 (0), 2243.2 (0),
2259.16 (0) }
[Click to access protein entry]
Spot: 2D-001DPN (dld1_human)
pI: 6.21 Mw: 58399
%vol: 0.0948838 %od: 0.347762
================
*TCPZ_HUMAN*
accession n°: P40227
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 904.507 (0), 916.513 (0), 937.51 (0), 1021.5 (0), 1076.58 (0),
1191.56 (0), 1255.71 (0), 1262.63 (0), 1498.78 (0), 1761.93 (0),
1768.06 (0), 1782.02 (0) }
Spot: 2D-001DPN (dld1_human)
pI: 6.21 Mw: 58399
%vol: 0.0948838 %od: 0.347762
================
*TCPZ_HUMAN*
accession n°: P40227
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 904.507 (0), 916.513 (0), 937.51 (0), 1021.5 (0), 1076.58 (0),
1191.56 (0), 1255.71 (0), 1262.63 (0), 1498.78 (0), 1761.93 (0),
1768.06 (0), 1782.02 (0) }
[Click to access protein entry]
Spot: 2D-001EOL (dld1_human)
pI: 5.36 Mw: 27132
%vol: 0.0935573 %od: 0.219983
================
*PHB_HUMAN*
accession n°: P35232
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1058.59 (0), 1149.68 (0), 1185.75 (0), 1213.85 (0), 1444.78 (0),
1478.88 (0), 1606.99 (0), 1998.27 (0) }
Spot: 2D-001EOL (dld1_human)
pI: 5.36 Mw: 27132
%vol: 0.0935573 %od: 0.219983
================
*PHB_HUMAN*
accession n°: P35232
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1058.59 (0), 1149.68 (0), 1185.75 (0), 1213.85 (0), 1444.78 (0),
1478.88 (0), 1606.99 (0), 1998.27 (0) }
[Click to access protein entry]
Spot: 2D-001EVT (dld1_human)
pI: 4.86 Mw: 22200
%vol: 0.0729592 %od: 0.166494
================
*TCTP_HUMAN*
accession n°: P13693
Identification Methods:
* MAPPING: {Im,Gm}
Spot: 2D-001EVT (dld1_human)
pI: 4.86 Mw: 22200
%vol: 0.0729592 %od: 0.166494
================
*TCTP_HUMAN*
accession n°: P13693
Identification Methods:
* MAPPING: {Im,Gm}
[Click to access protein entry]
Spot: 2D-001D1X (dld1_human)
pI: 6.79 Mw: 125156
%vol: 0.0479394 %od: 0.056564
================
*C1TC_HUMAN*
accession n°: P11586
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 878.455 (0), 940.493 (0), 983.569 (0), 1030.503 (0),
1097.601 (0), 1507.739 (0), 2239.078 (0) }
Spot: 2D-001D1X (dld1_human)
pI: 6.79 Mw: 125156
%vol: 0.0479394 %od: 0.056564
================
*C1TC_HUMAN*
accession n°: P11586
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 878.455 (0), 940.493 (0), 983.569 (0), 1030.503 (0),
1097.601 (0), 1507.739 (0), 2239.078 (0) }
[Click to access protein entry]
Spot: 2D-001DPP (dld1_human)
pI: 4.40 Mw: 58700
%vol: 0.0615732 %od: 0.118089
================
*CALR_HUMAN*
accession n°: P27797
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DPP (dld1_human)
pI: 4.40 Mw: 58700
%vol: 0.0615732 %od: 0.118089
================
*CALR_HUMAN*
accession n°: P27797
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DNM (dld1_human)
pI: 6.67 Mw: 60855
%vol: 0.0568198 %od: 0.106864
================
*HNRPL_HUMAN*
accession n°: P14866
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 923.431 (0), 978.479 (0), 1002.46 (0), 1076.62 (0), 1263.63 (0),
1881.85 (0) }
Spot: 2D-001DNM (dld1_human)
pI: 6.67 Mw: 60855
%vol: 0.0568198 %od: 0.106864
================
*HNRPL_HUMAN*
accession n°: P14866
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 923.431 (0), 978.479 (0), 1002.46 (0), 1076.62 (0), 1263.63 (0),
1881.85 (0) }
[Click to access protein entry]
Spot: 2D-001F8S (dld1_human)
pI: 4.93 Mw: 11055
%vol: 0.047534 %od: 0.0545988
================
*COX5A_HUMAN*
accession n°: P20674
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F8S (dld1_human)
pI: 4.93 Mw: 11055
%vol: 0.047534 %od: 0.0545988
================
*COX5A_HUMAN*
accession n°: P20674
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DXY (dld1_human)
pI: 6.78 Mw: 50698
%vol: 0.037106 %od: 0.0152738
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DXY (dld1_human)
pI: 6.78 Mw: 50698
%vol: 0.037106 %od: 0.0152738
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EUR (dld1_human)
pI: 6.46 Mw: 23087
%vol: 0.00630102 %od: 0.00304088
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EUR (dld1_human)
pI: 6.46 Mw: 23087
%vol: 0.00630102 %od: 0.00304088
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EEU (dld1_human)
pI: 5.73 Mw: 34283
%vol: 0.0140391 %od: 0.00531327
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EEU (dld1_human)
pI: 5.73 Mw: 34283
%vol: 0.0140391 %od: 0.00531327
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001FB3 (dld1_human)
pI: 5.35 Mw: 9218
%vol: 0.00718538 %od: 0.00879088
================
*ATP5J_HUMAN*
accession n°: P18859
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001FB3 (dld1_human)
pI: 5.35 Mw: 9218
%vol: 0.00718538 %od: 0.00879088
================
*ATP5J_HUMAN*
accession n°: P18859
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DV0 (dld1_human)
pI: 5.41 Mw: 53511
%vol: 0.0395748 %od: 0.0417547
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DV0 (dld1_human)
pI: 5.41 Mw: 53511
%vol: 0.0395748 %od: 0.0417547
================
*PDIA3_HUMAN*
accession n°: P30101
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EP6 (dld1_human)
pI: 4.60 Mw: 26759
%vol: 0.0486026 %od: 0.0718783
================
*1433S_HUMAN*
accession n°: P31947
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1008.798 (0), 1070.551 (0), 1205.655 (0), 1546.668 (0),
2189.874 (0), 2833.435 (0) }
Spot: 2D-001EP6 (dld1_human)
pI: 4.60 Mw: 26759
%vol: 0.0486026 %od: 0.0718783
================
*1433S_HUMAN*
accession n°: P31947
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1008.798 (0), 1070.551 (0), 1205.655 (0), 1546.668 (0),
2189.874 (0), 2833.435 (0) }
[Click to access protein entry]
Spot: 2D-001EJV (dld1_human)
pI: 5.47 Mw: 30658
%vol: 0.0937784 %od: 0.158736
================
*ANXA3_HUMAN*
accession n°: P12429
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 929.503 (0), 943.443 (0), 1018.52 (0), 1073.58 (0), 1091.62 (0),
1222.59 (0), 1350.67 (0), 1585.68 (0), 1673.88 (0), 1781.83 (0)
}
Spot: 2D-001EJV (dld1_human)
pI: 5.47 Mw: 30658
%vol: 0.0937784 %od: 0.158736
================
*ANXA3_HUMAN*
accession n°: P12429
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 929.503 (0), 943.443 (0), 1018.52 (0), 1073.58 (0), 1091.62 (0),
1222.59 (0), 1350.67 (0), 1585.68 (0), 1673.88 (0), 1781.83 (0)
}
[Click to access protein entry]
Spot: 2D-001E5O (dld1_human)
pI: 5.18 Mw: 41700
%vol: 0.094589 %od: 0.273344
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.464 (0), 1132.53 (0), 1198.7 (0), 1516.7 (0), 1790.87 (0),
1954.05 (0), 2231.07 (0) }
Spot: 2D-001E5O (dld1_human)
pI: 5.18 Mw: 41700
%vol: 0.094589 %od: 0.273344
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.464 (0), 1132.53 (0), 1198.7 (0), 1516.7 (0), 1790.87 (0),
1954.05 (0), 2231.07 (0) }
[Click to access protein entry]
Spot: 2D-001E1F (dld1_human)
pI: 6.35 Mw: 46694
%vol: 0.040275 %od: 0.0199574
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001E1F (dld1_human)
pI: 6.35 Mw: 46694
%vol: 0.040275 %od: 0.0199574
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001F8Z (dld1_human)
pI: 6.62 Mw: 10801
%vol: 0.0197137 %od: 0.0185161
================
*CX6A1_HUMAN*
accession n°: P12074
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F8Z (dld1_human)
pI: 6.62 Mw: 10801
%vol: 0.0197137 %od: 0.0185161
================
*CX6A1_HUMAN*
accession n°: P12074
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EK1 (dld1_human)
pI: 5.28 Mw: 30447
%vol: 0.0730698 %od: 0.131764
================
*TBB2C_HUMAN*
accession n°: P68371
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EK1 (dld1_human)
pI: 5.28 Mw: 30447
%vol: 0.0730698 %od: 0.131764
================
*TBB2C_HUMAN*
accession n°: P68371
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E13 (dld1_human)
pI: 6.81 Mw: 46815
%vol: 0.042449 %od: 0.0162818
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001E13 (dld1_human)
pI: 6.81 Mw: 46815
%vol: 0.042449 %od: 0.0162818
================
*M3K1_HUMAN*
accession n°: Q13233
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001F0H (dld1_human)
pI: 5.08 Mw: 17837
%vol: 0.0587359 %od: 0.0925391
================
*RM12_HUMAN*
accession n°: P52815
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.51 (0), 1155.619 (0), 1283.731 (0), 1360.731 (0)
}
Spot: 2D-001F0H (dld1_human)
pI: 5.08 Mw: 17837
%vol: 0.0587359 %od: 0.0925391
================
*RM12_HUMAN*
accession n°: P52815
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1045.51 (0), 1155.619 (0), 1283.731 (0), 1360.731 (0)
}
[Click to access protein entry]
Spot: 2D-001F7M (dld1_human)
pI: 6.90 Mw: 11989
%vol: 0.0437018 %od: 0.0338449
================
*HBB_HUMAN*
accession n°: P68871
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F7M (dld1_human)
pI: 6.90 Mw: 11989
%vol: 0.0437018 %od: 0.0338449
================
*HBB_HUMAN*
accession n°: P68871
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DRL (dld1_human)
pI: 5.21 Mw: 56625
%vol: 0.0874037 %od: 0.133725
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DRL (dld1_human)
pI: 5.21 Mw: 56625
%vol: 0.0874037 %od: 0.133725
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EEZ (dld1_human)
pI: 5.65 Mw: 34139
%vol: 0.0203033 %od: 0.00721078
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EEZ (dld1_human)
pI: 5.65 Mw: 34139
%vol: 0.0203033 %od: 0.00721078
================
*CRK_HUMAN*
accession n°: P46108
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DRH (dld1_human)
pI: 5.11 Mw: 56479
%vol: 0.0886934 %od: 0.155633
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DRH (dld1_human)
pI: 5.11 Mw: 56479
%vol: 0.0886934 %od: 0.155633
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001FA0 (dld1_human)
pI: 5.29 Mw: 10075
%vol: 0.0121967 %od: 0.00821031
================
*FABPL_HUMAN*
accession n°: P07148
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001FA0 (dld1_human)
pI: 5.29 Mw: 10075
%vol: 0.0121967 %od: 0.00821031
================
*FABPL_HUMAN*
accession n°: P07148
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EMI (dld1_human)
pI: 6.19 Mw: 28808
%vol: 0.0776758 %od: 0.122413
================
*PBLD_HUMAN*
accession n°: P30039
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EMI (dld1_human)
pI: 6.19 Mw: 28808
%vol: 0.0776758 %od: 0.122413
================
*PBLD_HUMAN*
accession n°: P30039
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DHC (dld1_human)
pI: 4.98 Mw: 75955
%vol: 0.0735488 %od: 0.0912603
================
*GRP78_HUMAN*
accession n°: P11021
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 986.574 (0), 997.61 (0), 1074.58 (0), 1191.65 (0), 1228.68 (0),
1329.6 (0), 1460.77 (0), 1566.79 (0), 1887.95 (0), 1934.01 (0),
1999.04 (0), 2164.92 (0) }
Spot: 2D-001DHC (dld1_human)
pI: 4.98 Mw: 75955
%vol: 0.0735488 %od: 0.0912603
================
*GRP78_HUMAN*
accession n°: P11021
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 986.574 (0), 997.61 (0), 1074.58 (0), 1191.65 (0), 1228.68 (0),
1329.6 (0), 1460.77 (0), 1566.79 (0), 1887.95 (0), 1934.01 (0),
1999.04 (0), 2164.92 (0) }
[Click to access protein entry]
Spot: 2D-001F5N (dld1_human)
pI: 5.40 Mw: 13625
%vol: 0.00788549 %od: 0.00761024
================
*TTHY_HUMAN*
accession n°: P02766
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F5N (dld1_human)
pI: 5.40 Mw: 13625
%vol: 0.00788549 %od: 0.00761024
================
*TTHY_HUMAN*
accession n°: P02766
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EM1 (dld1_human)
pI: 4.69 Mw: 28941
%vol: 0.0195663 %od: 0.0208181
================
*TPM3_HUMAN*
accession n°: P06753
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.451 (0), 940.463 (0), 1156.66 (0), 1243.66 (0), 1271.67 (0),
1284.75 (0), 1316.65 (0), 1642.8 (0), 1770.91 (0)
}
Spot: 2D-001EM1 (dld1_human)
pI: 4.69 Mw: 28941
%vol: 0.0195663 %od: 0.0208181
================
*TPM3_HUMAN*
accession n°: P06753
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 894.451 (0), 940.463 (0), 1156.66 (0), 1243.66 (0), 1271.67 (0),
1284.75 (0), 1316.65 (0), 1642.8 (0), 1770.91 (0)
}
[Click to access protein entry]
Spot: 2D-001DI0 (dld1_human)
pI: 5.16 Mw: 75169
%vol: 0.0134496 %od: 0.00531837
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DI0 (dld1_human)
pI: 5.16 Mw: 75169
%vol: 0.0134496 %od: 0.00531837
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EE9 (dld1_human)
pI: 6.30 Mw: 34500
%vol: 0.0692376 %od: 0.0456088
================
*LASP1_HUMAN*
accession n°: Q14847
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1067.6 (0), 1202.64 (0), 1418.76 (0), 1624.79 (0)
}
Spot: 2D-001EE9 (dld1_human)
pI: 6.30 Mw: 34500
%vol: 0.0692376 %od: 0.0456088
================
*LASP1_HUMAN*
accession n°: Q14847
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1067.6 (0), 1202.64 (0), 1418.76 (0), 1624.79 (0)
}
[Click to access protein entry]
Spot: 2D-001FBL (dld1_human)
pI: 6.85 Mw: 8761
%vol: 0.0682427 %od: 0.0706081
================
*UBC_HUMAN*
accession n°: P0CG48
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1039.35 (0), 1067.45 (0), 1081.39 (0) }
Spot: 2D-001FBL (dld1_human)
pI: 6.85 Mw: 8761
%vol: 0.0682427 %od: 0.0706081
================
*UBC_HUMAN*
accession n°: P0CG48
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1039.35 (0), 1067.45 (0), 1081.39 (0) }
[Click to access protein entry]
Spot: 2D-001EA8 (dld1_human)
pI: 5.81 Mw: 37445
%vol: 0.0357058 %od: 0.0342106
================
*ULAG_HUMAN*
accession n°: P31933
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EA8 (dld1_human)
pI: 5.81 Mw: 37445
%vol: 0.0357058 %od: 0.0342106
================
*ULAG_HUMAN*
accession n°: P31933
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001ERC (dld1_human)
pI: 6.28 Mw: 25260
%vol: 0.107891 %od: 0.230849
================
*TPIS_HUMAN*
accession n°: P60174
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1137.46 (0), 1234.54 (0), 1414.72 (0), 1458.64 (0), 1466.65 (0),
1586.701 (0), 1602.674 (0), 1614.754 (0), 1637.744 (0), 2191.964 (0),
3029.222 (0) }
Spot: 2D-001ERC (dld1_human)
pI: 6.28 Mw: 25260
%vol: 0.107891 %od: 0.230849
================
*TPIS_HUMAN*
accession n°: P60174
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1137.46 (0), 1234.54 (0), 1414.72 (0), 1458.64 (0), 1466.65 (0),
1586.701 (0), 1602.674 (0), 1614.754 (0), 1637.744 (0), 2191.964 (0),
3029.222 (0) }
[Click to access protein entry]
Spot: 2D-001EO3 (dld1_human)
pI: 5.50 Mw: 27320
%vol: 0.0744331 %od: 0.245937
================
*CATD_HUMAN*
accession n°: P07339
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EO3 (dld1_human)
pI: 5.50 Mw: 27320
%vol: 0.0744331 %od: 0.245937
================
*CATD_HUMAN*
accession n°: P07339
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DI1 (dld1_human)
pI: 5.25 Mw: 75561
%vol: 0.00814343 %od: 0.0015818
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DI1 (dld1_human)
pI: 5.25 Mw: 75561
%vol: 0.00814343 %od: 0.0015818
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DOJ (dld1_human)
pI: 6.04 Mw: 59281
%vol: 0.0766072 %od: 0.159768
================
*TCPG_HUMAN*
accession n°: P49368
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1098.65 (0), 1125.53 (0), 1166.72 (0), 1185.74 (0)
}
Spot: 2D-001DOJ (dld1_human)
pI: 6.04 Mw: 59281
%vol: 0.0766072 %od: 0.159768
================
*TCPG_HUMAN*
accession n°: P49368
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1098.65 (0), 1125.53 (0), 1166.72 (0), 1185.74 (0)
}
[Click to access protein entry]
Spot: 2D-001DKL (dld1_human)
pI: 6.63 Mw: 68813
%vol: 0.0472392 %od: 0.0492127
================
*PCKGM_HUMAN*
accession n°: Q16822
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 960.466 (0), 1000.59 (0), 1030.56 (0), 1079.6 (0), 1135.57 (0),
1271.7 (0), 1466.74 (0), 1725.93 (0), 1818.81 (0)
}
Spot: 2D-001DKL (dld1_human)
pI: 6.63 Mw: 68813
%vol: 0.0472392 %od: 0.0492127
================
*PCKGM_HUMAN*
accession n°: Q16822
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 960.466 (0), 1000.59 (0), 1030.56 (0), 1079.6 (0), 1135.57 (0),
1271.7 (0), 1466.74 (0), 1725.93 (0), 1818.81 (0)
}
[Click to access protein entry]
Spot: 2D-001E6E (dld1_human)
pI: 5.09 Mw: 41539
%vol: 0.0901673 %od: 0.211353
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.513 (0), 1132.57 (0), 1516.73 (0), 1790.89 (0), 1954.04 (0),
2231.05 (0) }
Spot: 2D-001E6E (dld1_human)
pI: 5.09 Mw: 41539
%vol: 0.0901673 %od: 0.211353
================
*ACTB_HUMAN*
accession n°: P60709
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 976.513 (0), 1132.57 (0), 1516.73 (0), 1790.89 (0), 1954.04 (0),
2231.05 (0) }
[Click to access protein entry]
Spot: 2D-001FAW (dld1_human)
pI: 5.71 Mw: 9362
%vol: 0.0403118 %od: 0.0236026
================
*TRFE_HUMAN*
accession n°: P02787
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001FAW (dld1_human)
pI: 5.71 Mw: 9362
%vol: 0.0403118 %od: 0.0236026
================
*TRFE_HUMAN*
accession n°: P02787
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EUK (dld1_human)
pI: 5.95 Mw: 23034
%vol: 0.086114 %od: 0.184042
================
*PRDX3_HUMAN*
accession n°: P30048
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EUK (dld1_human)
pI: 5.95 Mw: 23034
%vol: 0.086114 %od: 0.184042
================
*PRDX3_HUMAN*
accession n°: P30048
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DQZ (dld1_human)
pI: 5.16 Mw: 56190
%vol: 0.107707 %od: 0.779871
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DQZ (dld1_human)
pI: 5.16 Mw: 56190
%vol: 0.107707 %od: 0.779871
================
*CH60_HUMAN*
accession n°: P10809
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E4Y (dld1_human)
pI: 5.25 Mw: 43007
%vol: 0.0616469 %od: 0.101278
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.409 (0), 965.432 (0), 975.405 (0), 1041.56 (0), 1065.51 (0),
1267.6 (0), 1292.64 (0), 1319.64 (0), 1419.74 (0), 1522.78 (0)
}
Spot: 2D-001E4Y (dld1_human)
pI: 5.25 Mw: 43007
%vol: 0.0616469 %od: 0.101278
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.409 (0), 965.432 (0), 975.405 (0), 1041.56 (0), 1065.51 (0),
1267.6 (0), 1292.64 (0), 1319.64 (0), 1419.74 (0), 1522.78 (0)
}
[Click to access protein entry]
Spot: 2D-001DKJ (dld1_human)
pI: 5.54 Mw: 67048
%vol: 0.0856718 %od: 0.289076
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DKJ (dld1_human)
pI: 5.54 Mw: 67048
%vol: 0.0856718 %od: 0.289076
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001ECX (dld1_human)
pI: 4.97 Mw: 35604
%vol: 0.0371429 %od: 0.0208275
================
*HNRPC_HUMAN*
accession n°: P07910
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 903.376 (0), 943.584 (0), 1316.77 (0), 1329.66 (0)
}
Spot: 2D-001ECX (dld1_human)
pI: 4.97 Mw: 35604
%vol: 0.0371429 %od: 0.0208275
================
*HNRPC_HUMAN*
accession n°: P07910
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 903.376 (0), 943.584 (0), 1316.77 (0), 1329.66 (0)
}
[Click to access protein entry]
Spot: 2D-001DKQ (dld1_human)
pI: 5.47 Mw: 67397
%vol: 0.0760913 %od: 0.24099
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001DKQ (dld1_human)
pI: 5.47 Mw: 67397
%vol: 0.0760913 %od: 0.24099
================
*ALBU_HUMAN*
accession n°: P02768
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001E5D (dld1_human)
pI: 5.19 Mw: 43007
%vol: 0.0688322 %od: 0.0513565
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.514 (0), 965.479 (0), 975.47 (0), 1041.619 (0),
1065.577 (0), 1267.661 (0), 1292.688 (0), 1319.689 (0), 1419.755 (0),
1522.798 (0), 1884.034 (0) }
Spot: 2D-001E5D (dld1_human)
pI: 5.19 Mw: 43007
%vol: 0.0688322 %od: 0.0513565
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.514 (0), 965.479 (0), 975.47 (0), 1041.619 (0),
1065.577 (0), 1267.661 (0), 1292.688 (0), 1319.689 (0), 1419.755 (0),
1522.798 (0), 1884.034 (0) }
[Click to access protein entry]
Spot: 2D-001DTH (dld1_human)
pI: 4.77 Mw: 54763
%vol: 0.0872194 %od: 0.0803732
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
Spot: 2D-001DTH (dld1_human)
pI: 4.77 Mw: 54763
%vol: 0.0872194 %od: 0.0803732
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
[Click to access protein entry]
Spot: 2D-001EA1 (dld1_human)
pI: 5.65 Mw: 37445
%vol: 0.0690902 %od: 0.068037
================
*ULAG_HUMAN*
accession n°: P31933
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EA1 (dld1_human)
pI: 5.65 Mw: 37445
%vol: 0.0690902 %od: 0.068037
================
*ULAG_HUMAN*
accession n°: P31933
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DTP (dld1_human)
pI: 4.80 Mw: 54203
%vol: 0.0942574 %od: 0.0910386
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 910.467 (0), 966.572 (0), 1066.54 (0), 1081.67 (0), 1521.76 (0),
1780.83 (0), 1833.9 (0), 1965.03 (0) }
Spot: 2D-001DTP (dld1_human)
pI: 4.80 Mw: 54203
%vol: 0.0942574 %od: 0.0910386
================
*PDIA1_HUMAN*
accession n°: P07237
Identification Methods:
* MAPPING: {PMF,Gm}
peptide masses: { (TRYPSIN)
$m/Z= 910.467 (0), 966.572 (0), 1066.54 (0), 1081.67 (0), 1521.76 (0),
1780.83 (0), 1833.9 (0), 1965.03 (0) }
[Click to access protein entry]
Spot: 2D-001EUV (dld1_human)
pI: 5.15 Mw: 22770
%vol: 0.0537614 %od: 0.0948547
================
*APOA1_HUMAN*
accession n°: P02647
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001EUV (dld1_human)
pI: 5.15 Mw: 22770
%vol: 0.0537614 %od: 0.0948547
================
*APOA1_HUMAN*
accession n°: P02647
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001DIA (dld1_human)
pI: 5.21 Mw: 74392
%vol: 0.021851 %od: 0.00810027
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DIA (dld1_human)
pI: 5.21 Mw: 74392
%vol: 0.021851 %od: 0.00810027
================
*CD44_HUMAN*
accession n°: P16070
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001F7L (dld1_human)
pI: 6.87 Mw: 12036
%vol: 0.0455442 %od: 0.0417259
================
*HBB_HUMAN*
accession n°: P68871
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001F7L (dld1_human)
pI: 6.87 Mw: 12036
%vol: 0.0455442 %od: 0.0417259
================
*HBB_HUMAN*
accession n°: P68871
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001D9D (dld1_human)
pI: 6.74 Mw: 97476
%vol: 0.0613152 %od: 0.203748
================
*ACON_HUMAN*
accession n°: Q99798
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 935.521 (0), 1067.55 (0), 1170.66 (0), 1463.74 (0), 1601.78 (0),
1841.84 (0), 1861.97 (0) }
Spot: 2D-001D9D (dld1_human)
pI: 6.74 Mw: 97476
%vol: 0.0613152 %od: 0.203748
================
*ACON_HUMAN*
accession n°: Q99798
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 935.521 (0), 1067.55 (0), 1170.66 (0), 1463.74 (0), 1601.78 (0),
1841.84 (0), 1861.97 (0) }
[Click to access protein entry]
Spot: 2D-001EDQ (dld1_human)
pI: 4.77 Mw: 35011
%vol: 0.0376219 %od: 0.0157401
================
*NPM_HUMAN*
accession n°: P06748
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1568.72 (0), 1583.84 (0), 2160.96 (0), 2243.2 (0), 2259.17 (0)
}
Spot: 2D-001EDQ (dld1_human)
pI: 4.77 Mw: 35011
%vol: 0.0376219 %od: 0.0157401
================
*NPM_HUMAN*
accession n°: P06748
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1568.72 (0), 1583.84 (0), 2160.96 (0), 2243.2 (0), 2259.17 (0)
}
[Click to access protein entry]
Spot: 2D-001E1N (dld1_human)
pI: 5.40 Mw: 46098
%vol: 0.061131 %od: 0.0776987
================
*QCR1_HUMAN*
accession n°: P31930
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001E1N (dld1_human)
pI: 5.40 Mw: 46098
%vol: 0.061131 %od: 0.0776987
================
*QCR1_HUMAN*
accession n°: P31930
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001EXM (dld1_human)
pI: 4.91 Mw: 20813
%vol: 0.00762755 %od: 0.00381866
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EXM (dld1_human)
pI: 4.91 Mw: 20813
%vol: 0.00762755 %od: 0.00381866
================
*RRAS_HUMAN*
accession n°: P10301
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001EV9 (dld1_human)
pI: 6.86 Mw: 22876
%vol: 0.014076 %od: 0.00340819
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001EV9 (dld1_human)
pI: 6.86 Mw: 22876
%vol: 0.014076 %od: 0.00340819
================
*CDN1B_HUMAN*
accession n°: P46527
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001DXN (dld1_human)
pI: 6.23 Mw: 51091
%vol: 0.05822 %od: 0.0101196
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
Spot: 2D-001DXN (dld1_human)
pI: 6.23 Mw: 51091
%vol: 0.05822 %od: 0.0101196
================
*P53_HUMAN*
accession n°: P04637
Identification Methods:
* MAPPING: {Im}
[Click to access protein entry]
Spot: 2D-001E9G (dld1_human)
pI: 4.96 Mw: 38147
%vol: 0.0388379 %od: 0.0193946
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.525 (0), 1041.64 (0), 1122.56 (0), 1222.66 (0), 1554.74 (0),
1674.8 (0) }
Spot: 2D-001E9G (dld1_human)
pI: 4.96 Mw: 38147
%vol: 0.0388379 %od: 0.0193946
================
*K1C19_HUMAN*
accession n°: P08727
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 965.525 (0), 1041.64 (0), 1122.56 (0), 1222.66 (0), 1554.74 (0),
1674.8 (0) }
[Click to access protein entry]
Spot: 2D-001EWQ (dld1_human)
pI: 6.96 Mw: 21465
%vol: 0.0586622 %od: 0.143108
================
*PEBP1_HUMAN*
accession n°: P30086
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 885.495 (0), 937.458 (0), 1949.895 (0) }
Spot: 2D-001EWQ (dld1_human)
pI: 6.96 Mw: 21465
%vol: 0.0586622 %od: 0.143108
================
*PEBP1_HUMAN*
accession n°: P30086
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 885.495 (0), 937.458 (0), 1949.895 (0) }
[Click to access protein entry]
Spot: 2D-001ER2 (dld1_human)
pI: 6.17 Mw: 25202
%vol: 0.105865 %od: 0.446306
================
*PRDX6_HUMAN*
accession n°: P30041
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001ER2 (dld1_human)
pI: 6.17 Mw: 25202
%vol: 0.105865 %od: 0.446306
================
*PRDX6_HUMAN*
accession n°: P30041
Identification Methods:
* MAPPING: {Gm}
[Click to access protein entry]
Spot: 2D-001F3S (dld1_human)
pI: 7.06 Mw: 14905
%vol: 0.0987897 %od: 0.410652
================
*PPIA_HUMAN*
accession n°: P62937
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1055.51 (0), 1154.55 (0), 1310.53 (0), 1946.03 (0), 3306.74 (0)
}
Spot: 2D-001F3S (dld1_human)
pI: 7.06 Mw: 14905
%vol: 0.0987897 %od: 0.410652
================
*PPIA_HUMAN*
accession n°: P62937
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 1055.51 (0), 1154.55 (0), 1310.53 (0), 1946.03 (0), 3306.74 (0)
}
[Click to access protein entry]
Spot: 2D-001E58 (dld1_human)
pI: 5.15 Mw: 43228
%vol: 0.0502976 %od: 0.0235205
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.491 (0), 965.494 (0), 975.466 (0), 1041.629 (0),
1065.564 (0), 1292.675 (0), 1319.679 (0), 1419.739 (0), 1522.739 (0)
}
Spot: 2D-001E58 (dld1_human)
pI: 5.15 Mw: 43228
%vol: 0.0502976 %od: 0.0235205
================
*K1C18_HUMAN*
accession n°: P05783
Identification Methods:
* MAPPING: {PMF}
peptide masses: { (TRYPSIN)
$m/Z= 889.491 (0), 965.494 (0), 975.466 (0), 1041.629 (0),
1065.564 (0), 1292.675 (0), 1319.679 (0), 1419.739 (0), 1522.739 (0)
}
[Click to access protein entry]
Spot: 2D-001ENE (dld1_human)
pI: 4.50 Mw: 28086
%vol: 0.0278571 %od: 0.0289616
================
*EF1B_HUMAN*
accession n°: P24534
Identification Methods:
* MAPPING: {Gm}
Spot: 2D-001ENE (dld1_human)
pI: 4.50 Mw: 28086
%vol: 0.0278571 %od: 0.0289616
================
*EF1B_HUMAN*
accession n°: P24534
Identification Methods:
* MAPPING: {Gm}
SWISS-2DPAGE Viewer
|
Back to the |
-Get more information by dragging your mouse pointer over any highlighted spot -Click on a highlighted spot to access all its associated protein entries
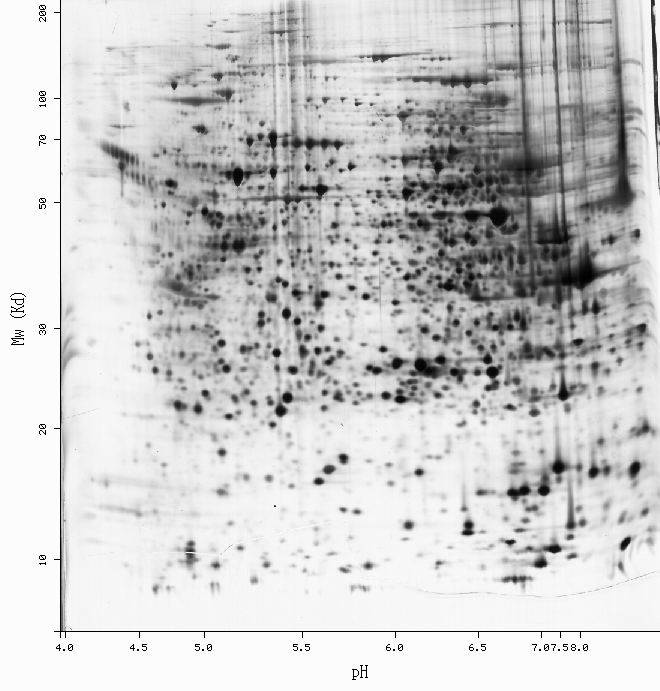
Back to the